Snp маркеры что это
ДНК маркеры в генеалогии
ДНК маркеры и для чего они нужны?
На чем основывается ДНК тест на этническое происхождение? На исследовании небольших участков ДНК, которые называются ДНК маркеры или локусы. ⠀
Существуют несколько разных типов ДНК маркеров, но для генеалогических исследований используются STR и SNP. ⠀
Простой тандемный повтор (от англ. Simple Tandem Repeats – STR) представляет собой короткую нуклеотидную последовательность, например GCAG, которая повторяется определенное количество раз: GCAG GCAG GCAG GCAG ⠀
В геноме человека насчитывается несколько десятков тысяч маркеров данного типа, но для установления этнического происхождения используется менее 200. ⠀
Однонуклеотидный полиморфизм (от англ. Single Nucleotide Polymorphism – SNP) это различия между двумя ДНК последовательностями по одному нуклеотиду. ⠀
Например, в одном случае последовательность может быть ATTTACGT, а в другом ACTTACGT. ⠀
В геноме человека имеется более 10 миллионов SNP маркеров. Для установления этнического происхождения используется менее 500 маркеров.
Каждый ДНК маркер имеет несколько вариантов, называемых аллельные варианты, или просто, аллели. ⠀
У STR маркеров количество повторов определенной нуклеотидной последовательности обозначает номер аллели. ⠀
Например, аллель 18 говорит о том, что по исследуемому маркеру наблюдается 18 повторов. ⠀
SNP маркеры, в большинстве случаев имеют только два аллельных варианта (в примере выше это будут аллели Т и С) хотя для некоторых маркеров количество аллелей может быть три, или даже четыре. ⠀
Если маркеры находятся на одной и той же молекуле ДНК, например хромосоме, или мтДНК, то совокупность наблюдаемых аллелей всех этих маркеров называется гаплотип ⠀
Группа схожих гаплотипов называется гаплогруппа и считается, что все индивидуумы, принадлежащие к одной гаплогруппе произошли от одного общего предка.
СОДЕРЖАНИЕ
SNP в кодирующей области бывают двух типов: синонимичные и несинонимичные SNP. Синонимичные SNP не влияют на последовательность белка, в то время как несинонимичные SNP изменяют аминокислотную последовательность белка.
Частота
В геноме
Геномное распределение SNP неоднородно; SNP встречаются в некодирующих областях чаще, чем в кодирующих областях или, в общем, там, где действует естественный отбор и «фиксирует» аллель (устраняя другие варианты) SNP, который составляет наиболее благоприятную генетическую адаптацию. Другие факторы, такие как генетическая рекомбинация и частота мутаций, также могут определять плотность SNP.
Внутри населения
Между популяциями людей существуют различия, поэтому аллель SNP, распространенный в одной географической или этнической группе, может быть намного реже в другой. Однако такая вариативность встречается относительно редко; в глобальной выборке из 67,3 миллионов SNP, проект Human Genome Diversity Project
не обнаружил таких частных вариантов, которые зафиксированы на данном континенте или в крупном регионе. Самые высокие частоты достигаются несколькими десятками вариантов, присутствующими в> 70% (и несколькими тысячами при> 50%) в Африке, Америке и Океании. Напротив, наиболее часто встречающиеся варианты, относящиеся к Европе, Восточной Азии, Ближнему Востоку или Центральной и Южной Азии, достигают всего от 10 до 30%.
Обладая этими знаниями, ученые разработали новые методы анализа популяционных структур у менее изученных видов. При использовании методов объединения стоимость анализа значительно снижается. Эти методы основаны на секвенировании популяции в объединенной выборке, а не на секвенировании каждого человека в популяции отдельно. С новыми инструментами биоинформатики появляется возможность исследовать структуру популяции, поток генов и миграцию генов, наблюдая частоты аллелей внутри всей популяции. Эти протоколы позволяют объединить преимущества SNP с микросателлитными маркерами. Однако при этом теряется информация, такая как неравновесие сцепления и информация о зиготности.
Приложения
Важность
Клинические исследования
Криминалистика
Некоторым недостатком использования SNP по сравнению с STR является то, что SNP предоставляют меньше информации, чем STR, и поэтому для анализа требуется больше SNP, прежде чем можно будет создать профиль подозреваемого. Кроме того, SNP во многом полагаются на наличие базы данных для сравнительного анализа образцов. Однако в случаях с деградированными или небольшими объемными выборками методы SNP являются отличной альтернативой методам STR. SNP (в отличие от STR) имеют множество потенциальных маркеров, могут быть полностью автоматизированы и позволяют сократить требуемую длину фрагмента до менее 100 пар оснований [26].
Фармакогенетика
Болезнь
Примеры
Базы данных
Как и для генов, существуют базы данных биоинформатики для SNP.
Рабочая группа International SNP Map картировала последовательность, фланкирующую каждый SNP, путем сопоставления с геномной последовательностью клонов с большими вставками в генбанке. Эти выравнивания были преобразованы в хромосомные координаты, которые показаны в таблице 1. Этот список значительно увеличился с тех пор, как, например, база данных Kaviar теперь перечисляет 162 миллиона однонуклеотидных вариантов (SNV).
| Хромосома | Длина (п. | Все SNP | SNP TSC | ||
|---|---|---|---|---|---|
| Всего SNP | кб на SNP | Всего SNP | кб на SNP | ||
| 1 | 214 066 000 | 129 931 | 1,65 | 75 166 | 2,85 |
| 2 | 222 889 000 | 103 664 | 2,15 | 76 985 | 2,90 |
| 3 | 186 938 000 | 93 140 | 2,01 | 63 669 | 2,94 |
| 4 | 169 035 000 | 84 426 | 2,00 | 65 719 | 2,57 |
| 5 | 170 954 000 | 117 882 | 1,45 | 63 545 | 2,69 |
| 6 | 165 022 000 | 96 317 | 1,71 | 53,797 | 3,07 |
| 7 | 149 414 000 | 71 752 | 2,08 | 42 327 | 3,53 |
| 8 | 125 148 000 | 57 834 | 2,16 | 42 653 | 2,93 |
| 9 | 107 440 000 | 62 013 | 1,73 | 43 020 | 2,50 |
| 10 | 127 894 000 | 61 298 | 2,09 | 42 466 | 3,01 |
| 11 | 129 193 000 | 84 663 | 1,53 | 47 621 | 2,71 |
| 12 | 125 198 000 | 59 245 | 2.11 | 38 136 | 3,28 |
| 13 | 93 711 000 | 53 093 | 1,77 | 35 745 | 2,62 |
| 14 | 89 344 000 | 44 112 | 2,03 | 29 746 | 3,00 |
| 15 | 73 467 000 | 37 814 | 1,94 | 26 524 | 2,77 |
| 16 | 74 037 000 | 38 735 | 1,91 | 23 328 | 3,17 |
| 17 | 73 367 000 | 34 621 | 2,12 | 19 396 | 3,78 |
| 18 | 73 078 000 | 45 135 | 1,62 | 27 028 | 2,70 |
| 19 | 56 044 000 | 25 676 | 2,18 | 11 185 | 5.01 |
| 20 | 63 317 000 | 29 478 | 2,15 | 17051 | 3,71 |
| 21 год | 33 824 000 | 20 916 | 1,62 | 9 103 | 3,72 |
| 22 | 33 786 000 | 28 410 | 1.19 | 11 056 | 3,06 |
| Икс | 131 245 000 | 34 842 | 3,77 | 20 400 | 6,43 |
| Y | 21 753 000 | 4 193 | 5,19 | 1,784 | 12,19 |
| RefSeq | 15,696 674 | 14 534 | 1.08 | ||
| Итоги | 2 710 164 000 | 1,419,190 | 1,91 | 887 450 | 3,05 |
Номенклатура
Номенклатура SNP включает несколько вариантов для отдельного SNP, но при этом отсутствует общий консенсус.
Общество вариации генома человека (HGVS) использует стандарт, который предоставляет дополнительную информацию о SNP. Примеры:
SNP анализ
SNP могут быть легко проанализированы из-за наличия только двух возможных аллелей и трех возможных генотипов, включающих два аллеля: гомозиготный A, гомозиготный B и гетерозиготный AB, что приводит к множеству возможных методов анализа. Некоторые включают: секвенирование ДНК ; капиллярный электрофорез ; масс-спектрометрия ; однонитевой конформационный полиморфизм (SSCP); одинарное расширение базы ; электрохимический анализ; денатурирующая ВЭЖХ и гель-электрофорез ; полиморфизм длины рестрикционного фрагмента ; и гибридизационный анализ.
Программы для прогнозирования эффектов SNP
Важной группой SNP являются те, которые соответствуют миссенс-мутациям, вызывающим изменение аминокислот на уровне белка. Точечная мутация конкретного остатка может по-разному влиять на функцию белка (от отсутствия эффекта до полного нарушения его функции). Обычно изменение аминокислот с аналогичным размером и физико-химическими свойствами (например, замена лейцина на валин) дает мягкий эффект и противоположный эффект. Точно так же, если SNP разрушает элементы вторичной структуры (например, замещение пролина в области альфа-спирали ), такая мутация обычно может повлиять на структуру и функцию всего белка. Используя эти простые и многие другие правила машинного обучения, была разработана группа программ для прогнозирования эффекта SNP:
Что такое SNP-исследование, STR и SNP маркеры?
Определение гаплотипа и гаплогруппы с помощью STR и SNP маркеров Y-хромосомы
Историческое происхождение народа, его происхождение и расселение может быть изучено разными методами. Среди них – проведение генетических тестов его представителей. Поскольку каждый народ представляет собой сообщество многочисленных родственников, то на роль объекта исследования наилучшим образом подходят мужчины, как наиболее социально активная часть этноса.
Среди генетических тестов есть те, что исследуют Y-хромосому (игрек-хромосому) и потому доступны только для мужчин. В них изучают Y-гаплотип (Y-STR, игрек-гаплотип) и Y-гаплогруппу (Y-SNP, игрек-гаплогруппа).
Y-гаплотип представляет собой совокупность маркёров, которая позволяет найти людей, которые являются друг другу близкими родственниками. Всего на игрек-хромосоме известно свыше 700 маркёров, но для исследований обычно используют только их часть.
Рассмотрим гаплотип, включающий в себя 18 маркёров хромосомы.:
Наименование маркёра говорит о его координате в игрек-хромосоме и повторяющегося фрагмента (мотива) цепочки азотистых оснований. В каждом маркёре расположены аллели, состоящие из тандемных повторов – повторяющихся комбинаций четырёх азотистых оснований (тетрануклеотид) – аденина (A), гуанина (G), цитозина (C), тимина (T).
Например, маркёр DYS393 располагается в позиции 3131152 прямой последовательности игрек-хромосомы (версия GRCh37/hg19). Его тетрануклеотид – AGAT (аденин-гуанин-аденин-тимин). В нашем примере DYS393=13. Это значит, что его повторяющийся мотив включает 13 тандемных повторов и выглядит следующим образом:
Различие между мужчинами может быть только в числе повторов. По современным данным, в локусе DYS393 наблюдается от 9 до 17 повторов (в нашем примере – 13).
Если у двух мужчин Y-гаплотипы совпадают, то это означает, что они родственники и происходят от одного предка.
Если совпадают 18 маркёров, то их предок жил от 1 до 250 поколений назад. Иначе говоря, это может быть как родной брат (или сын), так и человек, ближайший общий предок с которым жил более 6 тыс. лет назад. Как видим, диапазон очень широкий и лежит вне пределов непрерывного существования большинства современных народов. Чтобы сократить этот временной размах, нужно протестировать большее число маркёров – скажем, не 18, а 27. И если у двух мужчин совпадают 27 маркёров, то диапазон возраста ближайшего общего составляет уже от 1 до 160 поколений назад, т. е. в пределах последних 4 тыс. лет, что вписывается в рамки возникновения цивилизаций и народов.
Отсюда делаем вывод:
·
Чем больше маркёров совпадает, тем меньшее число поколений до общего предка;
·
Чем больше маркёров исследовано, тем меньше диапазон разброса.
Однако, для доказательства родства двух мужчин по прямой мужской линии одного только совпадения их Y-гаплотипов недостаточно. Также необходимо убедиться, совпадают ли их Y-гаплогруппы. Нередки случаи, что у двух мужчин совпадают 18 или даже 27 маркёров, но они относятся к разным гаплогруппам. Если не принимать во внимание их гаплогруппы, то может показаться, что они родственники. Явление, когда два внешне схожих гаплотипа относятся к разным гаплогруппам называется гомоплазия.
Гаплотип может использоваться для предварительного определения гаплогруппы. Для этой цели используют предикторы – специальные программы, которые предсказывают вашу гаплогруппу на основе значений маркёров. Достаточно ввести значения маркёров в программу и ваша гаплогруппа будет рассчитана с определённой степенью вероятности. Рассмотренный нами гаплотип относится к гаплогруппе R1a-M198 с вероятностью 85%. Определение гаплогруппы при помощи маркёров в большей степени результат статистической возможности, а не абсолютной определённости.
Поэтому, чтобы знать точно, к какому этносообществу принадлежит эта ДНК, необходимо провести исследование как гаплотипа (Y-STR
тест), так и гаплогруппы (Y-SNP
тест).
ДНК маркеры в генеалогии
ДНК маркеры и для чего они нужны?
На чем основывается ДНК тест на этническое происхождение? На исследовании небольших участков ДНК, которые называются ДНК маркеры или локусы. ⠀
Существуют несколько разных типов ДНК маркеров, но для генеалогических исследований используются STR и SNP. ⠀
Простой тандемный повтор (от англ. Simple Tandem Repeats – STR) представляет собой короткую нуклеотидную последовательность, например GCAG, которая повторяется определенное количество раз: GCAG GCAG GCAG GCAG ⠀
В геноме человека насчитывается несколько десятков тысяч маркеров данного типа, но для установления этнического происхождения используется менее 200. ⠀
Однонуклеотидный полиморфизм (от англ. Single Nucleotide Polymorphism – SNP) это различия между двумя ДНК последовательностями по одному нуклеотиду. ⠀
Например, в одном случае последовательность может быть ATTTACGT, а в другом ACTTACGT. ⠀
В геноме человека имеется более 10 миллионов SNP маркеров. Для установления этнического происхождения используется менее 500 маркеров.
Каждый ДНК маркер имеет несколько вариантов, называемых аллельные варианты, или просто, аллели. ⠀
У STR маркеров количество повторов определенной нуклеотидной последовательности обозначает номер аллели. ⠀
Например, аллель 18 говорит о том, что по исследуемому маркеру наблюдается 18 повторов. ⠀
SNP маркеры, в большинстве случаев имеют только два аллельных варианта (в примере выше это будут аллели Т и С) хотя для некоторых маркеров количество аллелей может быть три, или даже четыре. ⠀
Если маркеры находятся на одной и той же молекуле ДНК, например хромосоме, или мтДНК, то совокупность наблюдаемых аллелей всех этих маркеров называется гаплотип ⠀
Группа схожих гаплотипов называется гаплогруппа и считается, что все индивидуумы, принадлежащие к одной гаплогруппе произошли от одного общего предка.
СОДЕРЖАНИЕ
Методы на основе гибридизации
Динамическая аллель-специфическая гибридизация
Генотипирование с динамической аллель-специфической гибридизацией (DASH) использует преимущества различий в температуре плавления ДНК, которые возникают в результате нестабильности несовпадающих пар оснований. Этот процесс можно в значительной степени автоматизировать и включает в себя несколько простых принципов.
Поскольку генотипирование DASH измеряет количественное изменение Tm, оно способно измерять все типы мутаций, а не только SNP. Другие преимущества DASH включают возможность работы с датчиками без этикеток, а также простую конструкцию и рабочие условия.
Молекулярные маяки
Если последовательность зонда молекулярного маяка встречает свою геномную ДНК-мишень во время анализа, она отжигается и гибридизуется. Из-за длины последовательности зонда шпилька зонда будет денатурирована в пользу образования более длинного, более стабильного гибрида зонд-мишень. Это конформационное изменение позволяет флуорофору и тушителю быть свободными от их тесной близости из-за ассоциации шпильки, позволяя молекуле флуоресцировать.
Если, с другой стороны, последовательность зонда встречает последовательность-мишень всего с одним некомплементарным нуклеотидом, молекулярный маяк будет предпочтительно оставаться в своем естественном состоянии шпильки, и флуоресценция не будет наблюдаться, поскольку флуорофор остается погашенным.
Микромассивы SNP
Ферментные методы
Полиморфизма длин рестрикционных фрагментов
Полиморфизм длины рестрикционного фрагмента (RFLP) считается самым простым и ранним методом обнаружения SNP. SNP-RFLP использует множество различных эндонуклеаз рестрикции и их высокое сродство к уникальным и специфическим сайтам рестрикции. Выполняя расщепление геномного образца и определяя длину фрагментов с помощью гель-анализа, можно установить, разрезают ли ферменты ожидаемые сайты рестрикции. Неспособность разрезать геномный образец приводит к получению идентифицируемого более крупного, чем ожидалось, фрагмента, что означает наличие мутации в точке сайта рестрикции, которая обеспечивает защиту от нуклеазной активности.
К сожалению, сочетание факторов высокой сложности большинства эукариотических геномов, потребности в конкретных эндонуклеазах, того факта, что точная мутация не может быть обязательно разрешена в одном эксперименте, и медленный характер гель-анализов делают ПДРФ плохим выбором для высокой производительности. анализ.
Методы на основе ПЦР
Система ПЦР с устойчивой к амплификации тетра-праймером мутаций, или ARMS-PCR, использует две пары праймеров для амплификации двух аллелей в одной реакции ПЦР. Праймеры сконструированы таким образом, что две пары праймеров перекрываются в месте расположения SNP, но каждая идеально соответствует только одному из возможных SNP. Основа изобретения заключается в том, что неожиданно олигонуклеотиды с несовпадающим 3′-остатком не будут функционировать в качестве праймеров в ПЦР в соответствующих условиях. В результате, если данный аллель присутствует в реакции ПЦР, пара праймеров, специфичная для этого аллеля, будет производить продукт, но не для альтернативного аллеля с другим SNP. Две пары праймеров также сконструированы таким образом, что их продукты ПЦР имеют существенно разную длину, что позволяет легко различать полосы с помощью гель-электрофореза или анализа температуры плавления. При исследовании результатов, если геномный образец является гомозиготным, то продукты ПЦР, которые будут получены в результате, будут от праймера, который соответствует положению SNP, и внешнего праймера противоположной цепи, а также от двух внешних праймеров. Если геномный образец является гетерозиготным, то продукты будут происходить из праймера каждого аллеля и их соответствующих аналогов внешних праймеров, а также внешних праймеров.
Альтернативная стратегия заключается в проведении нескольких реакций кПЦР с разными наборами праймеров, нацеленных на каждый аллель отдельно. Хорошо сконструированные праймеры будут амплифицировать свой целевой SNP на гораздо более раннем цикле, чем другие SNP. Это позволяет различать более двух аллелей, хотя для каждого SNP требуется индивидуальная реакция qPCR. Для достижения достаточно высокой специфичности последовательность праймера может потребовать размещения искусственного несовпадения рядом с его 3′-концом, что является подходом, широко известным как Taq-MAMA.
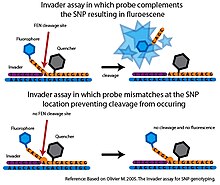
Эндонуклеаза лоскута
Удлинение грунтовки
Как правило, существует два основных подхода, в которых используется включение либо флуоресцентно меченных дидезоксинуклеотидов (ddNTP), либо флуоресцентно меченных дезоксинуклеотидов (dNTP). С ddNTP зонды гибридизуются с ДНК-мишенью непосредственно перед нуклеотидом SNP, и один ddNTP, комплементарный аллелю SNP, добавляется к 3′-концу зонда (отсутствующий 3′-гидроксил в дидиоксинуклеотиде предотвращает добавление дополнительных нуклеотидов. ). Каждый ddNTP помечен другим флуоресцентным сигналом, что позволяет обнаруживать все четыре аллеля в одной реакции. В случае dNTP аллель-специфические зонды имеют 3 ‘основания, комплементарные каждому из опрашиваемых аллелей SNP. Если целевая ДНК содержит аллель, комплементарный 3 ‘основанию зонда, целевая ДНК будет полностью гибридизоваться с зондом, позволяя ДНК-полимеразе распространяться от 3′ конца зонда. Это обнаруживается по включению флуоресцентно меченых дНТФ на конец зонда. Если целевая ДНК не содержит аллеля, комплементарного 3′-основанию зонда, целевая ДНК будет давать несоответствие на 3′-конце зонда, и ДНК-полимераза не сможет распространяться от 3′-конца зонда. Преимущество второго подхода состоит в том, что несколько меченых дНТФ могут включаться в растущую цепь, что способствует усилению сигнала. Однако в некоторых редких случаях ДНК-полимераза может происходить от несовпадающих 3’-зондов, давая ложноположительный результат.
Гибкость и специфичность удлинения праймера делают его пригодным для высокопроизводительного анализа. Зонды удлинения праймера могут быть размещены на предметных стеклах, что позволяет генотипировать сразу несколько SNP. Эта технология, широко известная как матричное удлинение праймеров (APEX), имеет несколько преимуществ по сравнению с методами, основанными на дифференциальной гибридизации зондов. Для сравнения, методы APEX обладают большей различающей способностью, чем методы, использующие эту дифференциальную гибридизацию, поскольку часто невозможно получить оптимальные условия гибридизации для тысяч зондов на ДНК-микрочипах (обычно это решается за счет наличия зондов с высокой избыточностью). Однако такая же плотность зондов не может быть достигнута в методах APEX, что приводит к снижению производительности за цикл.
Анализ Infinium компании Illumina Incorporated является примером конвейера полногеномного генотипирования, основанного на методе удлинения праймера. В анализе Infinium можно генотипировать более 100 000 SNP. В этом анализе используются нуклеотиды, меченные гаптеном, в реакции удлинения праймера. Гаптеновая метка распознается антителами, которые, в свою очередь, связаны с обнаруживаемым сигналом (Gunderson et al. 2006).
5′-нуклеаза
5′-нуклеазная активность ДНК-полимеразы Taq используется в анализе TaqMan для генотипирования SNP. Анализ TaqMan выполняется одновременно с реакцией ПЦР, и результаты можно считывать в режиме реального времени по мере протекания реакции ПЦР (McGuigan & Ralston 2002). Для анализа требуются прямые и обратные праймеры ПЦР, которые будут амплифицировать область, включающую полиморфный сайт SNP. Дискриминация аллелей достигается с помощью FRET в сочетании с одним или двумя аллель-специфическими зондами, которые гибридизуются с полиморфным сайтом SNP. Зонды будут иметь флуорофор, связанный с их 5 ‘концом, и молекулу гасителя, связанную с их 3′ концом. Пока зонд не поврежден, гаситель остается в непосредственной близости от флуорофора, устраняя сигнал флуорофора. На этапе амплификации ПЦР, если аллель-специфический зонд полностью комплементарен аллелю SNP, он будет связываться с цепью ДНК-мишени и затем разрушаться 5’-нуклеазной активностью полимеразы Taq, поскольку она расширяет ДНК из ПЦР. грунтовки. Разложение зонда приводит к отделению флуорофора от молекулы гасителя, генерируя обнаруживаемый сигнал. Если аллель-специфический зонд не является полностью комплементарным, он будет иметь более низкую температуру плавления и связываться не так эффективно. Это предотвращает действие нуклеазы на зонд (McGuigan & Ralston 2002).
Поскольку анализ TaqMan основан на ПЦР, его относительно просто реализовать. Анализ TaqMan можно объединить, объединив обнаружение до семи SNP в одной реакции. Однако, поскольку для каждого SNP требуется отдельный зонд, анализ TaqMan ограничен тем, насколько близко могут быть расположены эти SNP. Масштаб анализа можно резко увеличить, выполняя множество одновременных реакций в микротитровальных планшетах. Как правило, TaqMan ограничен приложениями, которые включают опрос небольшого количества SNP, поскольку для каждого SNP должны быть разработаны оптимальные зонды и условия реакции (Syvanen 2001).
Анализ лигирования олигонуклеотидов
ДНК-лигаза катализирует лигирование 3′-конца фрагмента ДНК с 5′-концом непосредственно прилегающего фрагмента ДНК. Этот механизм можно использовать для опроса SNP путем гибридизации двух зондов непосредственно над полиморфным сайтом SNP, в результате чего может происходить лигирование, если зонды идентичны целевой ДНК. В анализе олигонуклеотидной лигазы сконструированы два зонда; аллель-специфический зонд, который гибридизуется с целевой ДНК, так что его 3′-основание расположено непосредственно над нуклеотидом SNP, и второй зонд, который гибридизирует матрицу выше (ниже в комплементарной цепи) полиморфного сайта SNP, обеспечивая 5′-конец для реакции лигирования. Если аллель-специфический зонд совпадает с целевой ДНК, он полностью гибридизуется с целевой ДНК, и может произойти лигирование. Лигирование обычно не происходит в присутствии несоответствующего 3′-основания. Лигированные или нелигированные продукты могут быть обнаружены гель-электрофорезом, масс-спектрометрией MALDI-TOF или капиллярным электрофорезом для крупномасштабных приложений. С соответствующими последовательностями и тегами на олигонуклеотидах можно получить высокопроизводительные данные о последовательностях из лигированных продуктов и определенных генотипов (Curry et al., 2012). Использование большого количества индексов выборки позволяет сгенерировать данные последовательности с высокой пропускной способностью по сотням SNP в тысячах выборок за небольшую часть цикла высокопроизводительного секвенирования. Это массовое генотипирование с помощью технологии секвенирования (MGST).
Другие методы постамплификации, основанные на физических свойствах ДНК
Характерные свойства ДНК в виде температуры плавления и однонитевой конформации использовались в нескольких приложениях для различения аллелей SNP. Эти методы очень часто достигают высокой специфичности, но требуют высоко оптимизированных условий для получения наилучших возможных результатов.
Однонитевой конформационный полиморфизм
Одноцепочечная ДНК (оцДНК) складывается в третичную структуру. Конформация зависит от последовательности, и большинство мутаций одной пары оснований изменяют форму структуры. При нанесении на гель третичная форма будет определять подвижность оцДНК, обеспечивая механизм различения аллелей SNP. Этот метод сначала включает ПЦР-амплификацию целевой ДНК. Двухцепочечные продукты ПЦР денатурируют с использованием тепла и формальдегида для получения оцДНК. ОцДНК наносят на неденатурирующий гель для электрофореза и дают возможность свернуться в третичную структуру. Различия в последовательности ДНК изменяют третичную конформацию и обнаруживаются как различие в подвижности цепи оцДНК (Costabile et al. 2006). Этот метод широко используется, поскольку он технически прост, относительно недорог и требует использования общедоступного оборудования. Однако по сравнению с другими методами генотипирования SNP чувствительность этого анализа ниже. Было обнаружено, что конформация оцДНК сильно зависит от температуры, и, как правило, неясно, какова идеальная температура. Очень часто анализ будет проводиться с использованием нескольких различных температур. Также существует ограничение на длину фрагмента, потому что чувствительность падает, когда используются последовательности длиннее 400 п.н. (Costabile et al. 2006).
Гель-электрофорез в градиенте температуры
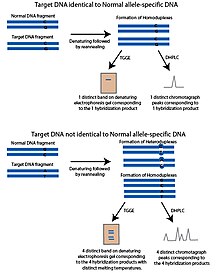
Метод гель-электрофореза в температурном градиенте (TGGE) или капиллярного электрофореза в температурном градиенте (TGCE) основан на том принципе, что частично денатурированная ДНК более ограничена и медленнее перемещается в пористом материале, таком как гель. Это свойство позволяет разделять ДНК по температуре плавления. Чтобы адаптировать эти методы для обнаружения SNP, используются два фрагмента; ДНК-мишень, которая содержит исследуемый полиморфный сайт SNP и аллель-специфичную последовательность ДНК, называемую нормальным фрагментом ДНК. Нормальный фрагмент идентичен целевой ДНК, за исключением потенциально полиморфного сайта SNP, который неизвестен в целевой ДНК. Фрагменты денатурируют, а затем повторно отжигают. Если целевая ДНК имеет тот же аллель, что и нормальный фрагмент, образуются гомодуплексы с такой же температурой плавления. При работе с гелем с градиентом температуры появится только одна полоса. Если целевая ДНК имеет отдельный аллель, после стадии повторного отжига образуются четыре продукта; гомодуплексы, состоящие из целевой ДНК, гомодуплексы, состоящие из нормальной ДНК, и два гетеродуплекса каждой цепи целевой ДНК, гибридизованные с нормальной цепью ДНК. Эти четыре продукта будут иметь разные температуры плавления и появятся в денатурирующем геле в виде четырех полос.
Денатурирующая высокоэффективная жидкостная хроматография
В денатурирующей высокоэффективной жидкостной хроматографии (DHPLC) используется обращенно-фазовая HPLC для исследования SNP. Ключом к DHPLC является твердая фаза, которая имеет дифференциальное сродство к одноцепочечной и двухцепочечной ДНК. В DHPLC фрагменты ДНК денатурируют нагреванием, а затем дают возможность повторно отожиться. Температура плавления повторно отожженных фрагментов ДНК определяет продолжительность времени, в течение которого они остаются в колонке. С помощью ПЦР генерируются два фрагмента; ДНК-мишень, содержащая полиморфный сайт SNP и аллель-специфичную последовательность ДНК, называемую нормальным фрагментом ДНК. Этот нормальный фрагмент идентичен целевой ДНК, за исключением потенциально полиморфного сайта SNP, который неизвестен в целевой ДНК. Фрагменты денатурируют, а затем дают возможность постепенно реанимировать. Реантированные продукты добавляют в колонку DHPLC. Если аллель SNP в целевой ДНК совпадает с нормальным фрагментом ДНК, только идентичные гомодуплексы будут формироваться на этапе повторного отжига. Если целевая ДНК содержит аллель SNP, отличный от нормального фрагмента ДНК, гетеродуплексы целевой ДНК и нормальной ДНК, содержащие несовпадающий полиморфный сайт, будут формироваться в дополнение к гомодуплексам. Несоответствующие гетеродуплексы будут иметь температуру плавления, отличную от температуры гомодуплексов, и не будут удерживаться в колонке так долго. Это создает образец хроматографии, который отличается от образца, который был бы сгенерирован, если бы целевой фрагмент ДНК и нормальные фрагменты ДНК были идентичны. Элюированную ДНК детектируют по УФ-поглощению.
DHPLC легко автоматизировать, так как не требуется мечение или очистка фрагментов ДНК. Метод также относительно быстр и отличается высокой специфичностью. Одним из основных недостатков DHPLC является то, что температура колонки должна быть оптимизирована для каждой мишени, чтобы достичь нужной степени денатурации.
Плавление всего ампликона с высоким разрешением
Сканирование больших ампликонов основано на тех же принципах, что описаны выше. Однако информативными становятся температура плавления и общая форма кривой плавления. Для ампликонов размером> 150 п.н. часто имеется> 2 пика плавления, каждый из которых может варьироваться в зависимости от состава матрицы ДНК. Многочисленные исследователи смогли успешно устранить большую часть своего секвенирования с помощью сканирования на основе расплава, что позволило точно определить генотипирование на основе локуса большого числа людей. Многие исследователи обнаружили, что сканирование мутаций с использованием плавления с высоким разрешением является жизнеспособным и практичным способом изучения целых генов.
Использование белков, связывающих несовпадение ДНК
Белки, связывающие ошибочные спаривания ДНК, могут различать одиночные нуклеотидные несовпадения и, таким образом, облегчают дифференциальный анализ SNP. Например, белок MutS из Thermus aquaticus связывает разные однонуклеотидные несовпадения с разным сродством и может использоваться в капиллярном электрофорезе для дифференциации всех шести наборов несовпадений (Drabovich & Krylov 2006).
SNPlex
Сюрвейерский анализ нуклеазы
Нуклеаза-сюрвейер представляет собой фермент эндонуклеазы несовпадения, который распознает все замены оснований и небольшие вставки / делеции (инделки) и расщепляет 3′-сторону несовпадающих сайтов в обеих цепях ДНК.