Rna seq что это
Секвенирование единичных клеток (версия — Metazoa)
Кит секвенирования стремительно плывет в страну Единичной клетки
Автор
Редакторы
Статья на конкурс «био/мол/текст»: Ученые исследовали единичные клетки со времен изобретения микроскопа. Но такие методы-киты молекулярной биологии, как анализ на микрочипах, секвенирование нуклеиновых кислот и масс-спектрометрия подразумевают использование некоторой массы клеток. Однако если взять среднее большого числа клеток, то на выходе получим усредненный результат. Есть области, где усредненный результат — это уже неинтересно; есть важные фундаментальные вопросы, на которые вам ответит только она — Единичная клетка. Например, этот подход может стать одним из ключей к познанию тайн эмбрионального развития, закономерностей дифференциации стволовых клеток, функционирования нейронов и выработке стратегий лечения опухолевых заболеваний.
Конкурс «био/мол/текст»-2013
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2013 в номинации «Лучший обзор».
Спонсор конкурса — дальновидная компания Thermo Fisher Scientific. Спонсор приза зрительских симпатий — фирма Helicon.
Об эпигенетике и ее роли в реализации генетической программы клетки читайте в статье «Развитие и эпигенетика, или история о минотавре». — Ред.
Чужой среди чужих
Если нормальные клетки организма в большинстве своем имеют идентичную генетическую информацию, то опухолевые клетки по определению «чужие». Более того, клетки одной опухоли (за редким исключением) различаются между собой по набору генетических аберраций. Поэтому в результате исследования образцов злокачественной ткани мы получаем результат, характеризующий большинство клеток. Применяем терапию, направленную на обнаруженные мишени, и убиваем в лучшем случае это большинство. А тем временем меньшая часть клеток, не обладающая этими мутациями, может выжить [5]. В этом случае высок риск рецидива. Вот здесь могло бы прийти на помощь секвенирование ДНК единичных клеток. Кстати, исследуя единичные клетки опухоли, можно узнать ее эволюцию. Так, в результате секвенирования ста геномов единичных клеток двух образцов рака молочной железы подтвердилось, что метастазирующие клетки возникают на поздних этапах развития опухоли [6]. Кроме того, был обнаружен паттерн роста опухоли, который они назвали «точечной клональной эволюцией», заимствовав термин из теории эволюции видов. При этом опухолевые популяции отличались от их предка, без очевидного промежуточного ветвления. То есть скорость геномной эволюции значительно превышала эффективный рост популяции.
Единичных клеток будет достаточно для неинвазивного мониторинга за заболеванием (циркулирующие опухолевые клетки, воздействие химиотерапии) [7]. В будущем это может быть расширено, например, на вирусные заболевания и процесс их мутагенеза в зараженных популяциях. Интересно применение методов секвенирования единичных клеток для изучения редких образцов, таких как палеонтологические, — например, из животных, заключенных в янтарь, которые обычно очень маленького размера.
Суммируя вышесказанное, цели секвенирования единичных клеток многоклеточных организмов разнообразны и затрагивают многие актуальные вопросы биологии. Каковы технические особенности этой процедуры?
Достать любой ценой
Секвенирование нуклеиновых кислот — это определение последовательности нуклеотидов в молекулах ДНК или РНК. Общая схема исследования единичных клеток секвенированием выглядит так: получение единичных клеток, выделение нуклеиновых кислот, подготовка библиотеки, собственно секвенирование и биоинформатический анализ. Кроме того, нужно не забывать про систему контролей [8]. Несмотря на то, что каждая клетка уникальна и полученные результаты не воспроизвести, исследования необходимо проводить параллельно с отрицательным контрольным образцом без клетки и положительным контрольным образцом нуклеиновых кислот с концентрацией, сопоставимой с таковой в экспериментальном образце.
Получить единичные клетки можно разными методами. Не потребует особого инструментария метод лимитирующих разведений. Однако при работе с тканями будет необходимо подбирать оптимальные условия для диссоциации клеток. При наличии микроскопа можно научиться захватывать единичные клетки пипеткой. Этот прием использовал пионер секвенирования единичных клеток Джеймс Эбервайн (James Eberwine) [9]. Он подойдет для исследования крупных клеток, — например, нейронов [10] или яйцеклеток. Можно попытаться даже исследовать отдельные компартменты клетки (в случае нейрона — дендриты). Именно так был обнаружен новый класс транскриптов — цитоплазматические транскрипты, сохраняющие интронную последовательность (cytoplasmic intron-sequence retaining transcripts), которые, по-видимому, участвуют в посттранскрипционной регуляции экспрессии генов [11].
При наличии инструментов, опыта и мастерства микроскописта лазерная микродиссекция открывает возможность получения единичных клеток и их компартментов из любых тканей [12]. Для выделения единичных клеток (а также ядер или даже отдельных хромосом) из жидких тканей или суспензий клеток отлично подойдет проточная цитометрия [13]. Одним из перспективных методов автоматизации получения единичных клеток является микрофлуидика. Из введенной в чип массы клеток с помощью специальных ловушек отсеиваются отдельные клетки. Так, на основе технологии микрофлуидики разработано устройство для сепарации хромосом [14]. Это позволяет получить информацию о гаплотипе без исследования семейного трио. (Гаплотип — это совокупность материнских и отцовских аллелей на локусах одной хромосомы; его необходимо знать для интерпретации выявленных мутаций.) Есть возможность на микрофлуидном чипе провести операции выделения РНК, обратной транскрипции и преамплификации. Это упрощает, ускоряет и унифицирует процесс, предшествующий приготовлению библиотек.
Таким образом, методов получения единичных клеток много, и остается подобрать оптимальный в соответствии с целями, умениями и инструментами.
Приумножаем богатство
Дальше из полученных единичных клеток нужно выделить нуклеиновые кислоты. Типичная клетка содержит меньше сотни пикограмм ДНК и около 10 пг тотальной РНК (мРНК обычно составляет примерно 1–5% от тотальной РНК). Этого количества недостаточно для приготовления библиотек с помощью коммерческих наборов, где в среднем требуется 100 нг ДНК для полногеномного секвенирования, 100 нг РНК для секвенирования транскриптома (в англоязычной литературе — RNA-seq). Отсюда два вывода. Во-первых, при выделении нуклеиновых кислот, как отдельной процедуре, можно запросто потерять весь материал. Поэтому чаще всего проводят просто лизис клетки в пробирке и в ней же проводят последующие процедуры. Во-вторых, это означает, что придется иметь дело с полногеномной амплификацией (whole genome amplification — WGA) или амплификацией транскриптома. Рассмотрим сначала методы амплификации ДНК.
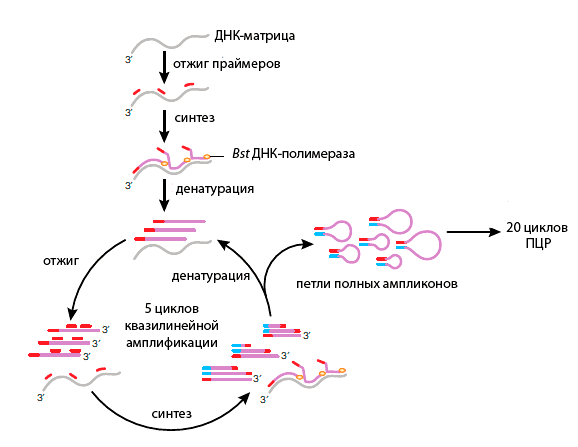
Более 10 лет самым надежным методом WGA считался метод амплификации со множественным замещением цепи (multiple displacement amplification — MDA) [15]. В этой реакции случайные праймеры удлиняются с помощью фермента с выраженной способностью к замещению цепи — phi29 ДНК-полимеразой. В отличие от амплификации с помощью ПЦР, точность прочтения с помощью MDA на 3–4 порядка выше, и ампликоны имеют среднюю длину >12 т.п.н. Несмотря на высокую точность этой полимеразы, применение MDA для амплификации ДНК единичных клеток показало, что часто не удается определить аллель: гетерозигота ошибочно считается гомозиготой. Но теперь разработан новый метод полногеномной амплификации, подходящий для единичных клеток — MALBAC (Multiple annealing and looping-based amplification cycles) [16]. Он соединяет в себе черты ПЦР и MDA, что приводит к снижению ошибки амплификации (рис. 1). В этом методе используются две ДНК-полимеразы — Bst и Taq. Однако и MALBAC не лишен недостатков: при генотипировании вариантов единичных нуклеотидов этот метод дает много ложноположительных результатов. После амплификации можно приступать к подготовке полногеномной или экзомной библиотек с помощью обычных коммерческих наборов.
Рисунок 1. Упрощенная схема реакции MALBAC. Праймеры MALBAC (красные) имеют общую последовательность и 8 случайных нуклеотидов, которые отжигаются на ДНК-матрице. Синтез с замещением цепи приводит к образованию частичных ампликонов, которые затем денатурируют. Отжиг на новых позициях ДНК-матрицы увеличивает покрытие генома, а ошибок амплификации в результате немного. Отжиг праймеров на частичных ампликонах с последующим синтезом приводит к образованию полных ампликонов. Они имеют на 5′-конце последовательность праймера MALBAC (красная), а на 3′-конце комплиментарную ему последовательность (голубая). Денатурация приводит к регенерации исходной матрицы и образованию еще более разнообразного набора частичных ампликонов. Полные ампликоны образуют петли, которые могут быть устойчивы к последующей амплификации и гибридизации. Полные ампликоны генерируются в течение пяти циклов и затем амплифицируются экспоненциально в ходе 20 циклов ПЦР с использованием праймеров к общему участку праймеров MALBAC. Bst — Bacillus stearothermophilus.
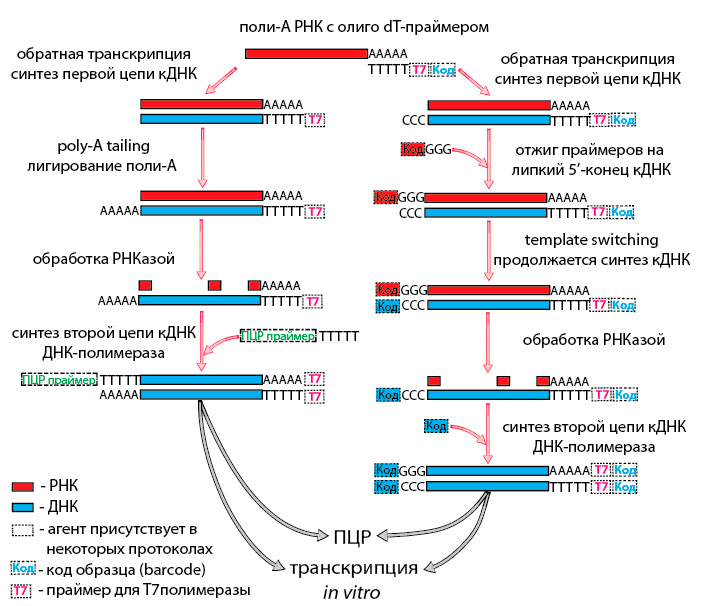
Теперь обратимся к амплификации транскриптома единичной клетки. Не будем забывать, что РНК плавает в лизате. Нацеленно воздействовать на нее в таком окружении пока возможно только через поли-А хвост, который является характерным признаком зрелых мРНК. Следовательно, пока на уровне транскриптома единичных клеток нам недоступен весь остальной мир РНК. Итак, олиго-dT праймер на поли-А хвосте, синтезируем первую цепь кДНК. А далее есть несколько путей, как получить универсальный «якорь», за который можно будет манипулировать кДНК во всех последующих операциях. Можно выделить два основных приема: poly-A tailing и template switching (рис. 2).
Рисунок 2. Общая схема методов RNA-Seq (пояснения в тексте)
В первом случае к первой цепи кДНК лигируют поли-А хвост — он будет «якорем» [18], [19]. А template switching — переключение матрицы — это свойство РНК-зависимой ДНК-полимеразы: дойдя до 5′-конца матрицы, она присоединяет к кДНК еще несколько оснований без нее, и получается «липкий» конец. К этому концу (как правило, 3C) присоединяются специальные праймеры с 3G на 5′-конце. РНК-зависимая ДНК-полимераза переключается, и теперь праймер служит ей матрицей для синтеза кДНК. Комплементарный праймеру участок первой цепи кДНК и будет «якорем» [20]. После создания «якоря» синтезируют вторую цепь кДНК и приступают к амплификации. Это можно сделать с помощью ПЦР [19], [20] или с помощью транскрипции в пробирке (in vitro transcription — IVT); ее также называют синтезом аРНК [21], [22]. Преимуществом ПЦР является высокая степень амплификации (10 6 ) и высокая скорость реакции, но надо учитывать наличие характерных для этой реакции ошибок и побочных неспецифических продуктов.
Методы, в которых РНК нарабатывается с помощью IVT, имеют высокую точность, но они длительные. Кроме того, степень амплификации при IVT довольно низка (10 3 ), что требует нескольких раундов этого процесса. Существует также возможность объединить несколько образцов в одной реакционной пробирке для приготовления библиотеки. Для этого при синтезе кДНК каждому образцу присваивается уникальный код (barcode) в составе одного из праймеров [21], [23]. Однако при таком подходе теряется возможность прочесть всю последовательность транскриптов, так как на стадии приготовления библиотек кДНК или РНК фрагментируют, и с кодом остаются лишь 3′- или 5′-концы транскриптов. Вообще методов амплификации транскриптома много и они плавно перетекают друг в друга. Полученную после амплификации кДНК или РНК используют для приготовления библиотеки и последующего секвенирования с помощью коммерчески доступных наборов [24].
Опасные последствия
Рисунок 3. Образное изображение захлестывающего количества информации, генерируемой в результате секвенирования нового поколения (next generation sequencing — NGS).
иллюстрация Артура Рэкама к книге Л. Кэрролла «Алиса в стране чудес»; автор идеи Бабенко В.В.
Когда мы думаем о секвенировании, нельзя ни в коем случае забывать о биоинформатическом анализе получаемой на выходе сырой информации, иначе можно не получить ожидаемой отдачи от этого метода исследования (рис. 3). Здесь сложно выделить универсальный рецепт, каждое исследование будет иметь свои особенности и трудности. Например, необходимо учитывать стохастическую экспрессию генов в генетически идентичных клетках [25], [26].
Взгляд в будущее
Одной из главных целей биологии является понимание связи между генетическим материалом, генетическими регуляторными сетями и фенотипом индивидуальной клетки (в физиологическом или патологическом состоянии). Исходя из этого, ясны пути дальнейшего развития технологии секвенирования единичных клеток. Во-первых, научиться на уровне одной клетки изучать все разнообразие РНК, а не только поли-А РНК; сохранять информацию о принадлежности к цепи (смысловой или несмысловой); секвенировать транслирующиеся РНК. Во-вторых, иметь возможность одновременно секвенировать геном, эпигеном и транскриптом одной клетки. Наконец, чтобы приблизиться к объяснению того, как клетка выбирает путь развития, как регулируется дифференциация, нужно научиться комбинировать секвенирование РНК и ДНК с визуализацией живых клеток с мечеными белками [27].
Кит секвенирования единичных клеток еще только набирает мили, так что сейчас есть возможность запрыгнуть ему на спину и проложить маршрут в неизведанные океаны. Чего вам и желаю!
Малая РНК большого значения
Малая РНК большого значения
Малая некодирующая РНК сальмонелл влияет не только на экспрессию бактериальных генов, но и на экспрессию генов клетки хозяйского организма.
Автор
Редакторы
Параллельный анализ молекул РНК, которые синтезируются сальмонеллами и клетками человека во время инфекции, показал, как бактериальная малая РНК изменяет транскрипционные профили не только клеток сальмонеллы, но и клеток человека.
Что происходит, когда бактерия сталкивается с клетками хозяйского организма? А когда проникает внутрь? За счет чего бактерия выживает в новой для нее среде? Как клетки хозяина защищаются от патогенов? Чтобы ответить на все эти вопросы, необходимо понять, как изменяется транскрипция генов во взаимодействующих клетках. За последние годы было разработано множество подходов: от анализа экспрессии генов in vivo до секвенирования полного транскриптома бактерий и хозяйских клеток. Однако все они фокусировались на матричных РНК одного из объектов (бактерии или клетки-хозяина), но не обоих сразу [1].
Полногеномное секвенирование РНК (RNA-seq) — это очень чувствительный метод глобального анализа экспрессии генов, использующийся в инфекционной биологии. Однако в изучении внутриклеточных бактериальных инфекций есть свои трудности: в процессе участвуют два разных организма с совершенно разными наборами РНК. Обычно эти организмы физически разделяют, а затем секвенируют транскриптомы каждого из них. Причем ограничиваются лишь матричными РНК, поскольку именно они дают информацию о производимых в клетке белках, и пренебрегают РНК, считывающимися с некодирующих регионов [2].
RNA-seq
RNA-seq — это метод высокопроизводительного секвенирования (или секвенирования нового поколения, NGS) полного транскриптома, позволяющий определять последовательность РНК с высокой точностью (до 99,9%) и производить параллельно до 500 000 процессов секвенирования [3–5].
Определение последовательности РНК производится либо секвенированием кДНК, либо прямым секвенированием фрагментов РНК. Сейчас для этого используют несколько технологических платформ:
Бактерии синтезируют множество некодирующих малых РНК (small RNA, sRNA) [7], роль которых в патогенезе всё еще остается малопонятной — ведь такие РНК не находят своего отражения в фенотипе вирулентности. Эти малые РНК оказывают посттранскрипционное влияние на экспрессию генов у многих организмов. Исключением не является и Salmonella enterica серовар Typhimurium, у которой транскрибируются сотни малых регуляторных РНК. Многие из них активны лишь в условиях стресса или при инфицировании. Однако делеция генов таких РНК приводит к незначительному изменению фенотипа — возможно, потому, что малые РНК отвечают за точную настройку процессов экспрессии, а не за сами процессы. Чтобы узнать функции малых РНК во время инфекции, необходимы высокочувствительные методы исследования.
Salmonella enterica subsp. enterica, серовар Typhimurium
Клетки S. enterica sv. Typhimurium.
Сальмонеллы — грамотрицательные палочковидные бактерии. Salmonella enterica — один из двух видов сальмонелл, насчитывающий 6 подвидов и более 2500 сероваров. Эти организмы — факультативные анаэробы, вызывающие преимущественно желудочно-кишечные заболевания: сальмонеллёз, брюшной тиф, паратифы.
При попадании в организм человека Salmonella Typhimurium развивается сальмонеллёз (брюшной тиф вызывает Salmonella Typhi). Этот серовар — не единственный возбудитель сальмонеллёза, но в большинстве внутрибольничных (нозокомиальных) вариантов болезни повинен именно он [8]. Вне стационаров обычно заражаются при поедании сырых/недоваренных яиц, реже — молочных и мясных продуктов, опять же, не прошедших должную термическую обработку. А можно просто не помыть руки или кухонный инвентарь после работ со всеми этими продуктами — и неделя лихорадки и пробежек до санузла гарантирована.
Оказавшись в кишечнике, сальмонеллы стремятся попасть в собственную пластинку слизистой оболочки. Потому кишечный эпителий для них — барьер, который бактерии должны миновать. Для этого сальмонеллы провоцируют перестройку цитоскелета клеток эпителия, приводящую к образованию складок в их мембране. Процесс контролируется двухкомпонентной сигнальной системой PhoP/PhoQ, регулирующей экспрессию генов вирулентности, и приводит к заключению бактерий в эндосомы, в которых они переносятся через эпителиальные клетки в собственную пластинку слизистой [9], [10]. Стоит отметить, что сальмонеллы «болтливы» и решают сложные задачи коллективно: некоторые гены вирулентности находятся у них под контролем чувства кворума [11]. Во время инвазии сальмонеллы транскрибируют гены из региона, называемого островом патогенности Salmonella 1 (Salmonella pathogenicity island 1, SPI-1). Когда бактерии оказываются внутри клетки, активность этого острова падает и начинается транскрипция генов из региона SPI-2, которые отвечают за внутриклеточную выживаемость [1].
S. Typhimurium — вообще очень хитрая бактерия, нашпигованная крайне полезными мобильными генетическими элементами разного происхождения — как профагами, кодирующими ферменты конверсии поверхностных антигенов сальмонеллы и факторы вирулентности, так и геномными островами, предоставляющими бактерии-хозяину не только факторы патогенности, но и устойчивость к антибиотикам.
Группа ученых из нескольких институтов Германии предложила новый метод изучения РНК клеток человека и их внутренних паразитов — бактерий [2]. Исследователи решили провести одновременный анализ РНК патогена и хозяйской клетки — двойное секвенирование РНК (dual RNA-seq). Причем не стали ограничиваться лишь матричными РНК, а изучили и малые регуляторные РНК бактерий.
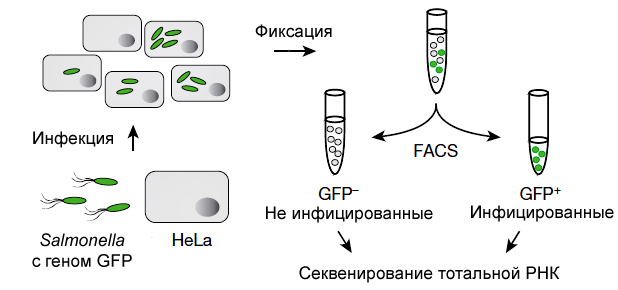
Эксперимент с человеческими клетками, инфицированными сальмонеллой
А. Вестерманн и коллеги использовали Salmonella Typhimurium и клетки линии HeLa, в которых бактерия может длительно размножаться [2]. Идентификацию клеток HeLa с сальмонеллами внутри проводили по зеленому флуоресцентному белку (GFP), синтезируемому бактериями. Методом флуоресцентно-активированного отбора (Fluorescence Activated Cell Sorting, FACS) изучали клетки на 4-м часу после инфицирования (в среднем там находили 10 бактерий) и по прошествии 24 часов (75 бактерий). В качестве контроля использовали неинфицированные клетки (рис. 1). Из образцов выделяли тотальную РНК и на аппаратуре Illumina проводили двойное секвенирование кодирующих и некодирующих молекул.
Рисунок 1. Схема двойного секвенирования РНК (dual RNA-seq).
Малые РНК внутриклеточных сальмонелл
Чтобы повысить информативность двойного секвенирования, исследователи проанализировали малые РНК бактерий в образцах, взятых не только через 4 и 24 часа, но и через 2, 8 и 16 часов после инфицирования. Таким образом были идентифицированы 145 известных и 189 новых малых РНК сальмонеллы. Синтез некоторых из них возрастал более чем в 10 раз уже на второй час после инвазии.
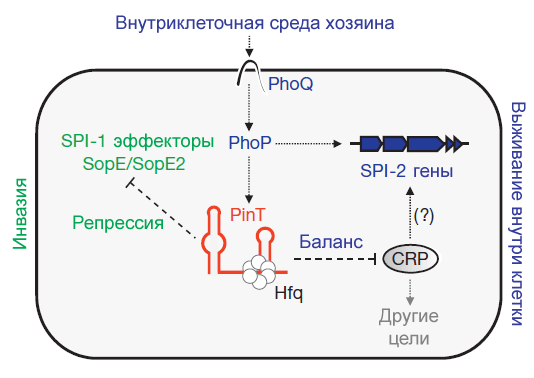
Функция малых РНК состоит в том, что, соединяясь с целевыми матричными РНК, они модулируют их стабильность и экспрессию. Исследователи обнаружили наиболее активную 80-нуклеотидную малую РНК, чья транскрипция возрастала более чем в 100 раз во время инфекции, и назвали ее PinT. Ген этой РНК приобретен сальмонеллами горизонтально и активируется бактериальной сигнальной системой PhoP/PhoQ.
Анализ показал, что PinT снижает уровень транскрипции генов sopE и sopE2 из региона SPI-1 — генов эффекторных белков, которые участвуют в процессе инвазии (рис. 2). Также эта малая РНК регулирует экспрессию РНК белка CRP (рецептора цАМФ), который участвует в активации генов внутриклеточной выживаемости бактерий. Блокируя РНК CRP, PinT модулирует активность второго острова патогенности — SPI-2.
Рисунок 2. Схема регуляции экспрессии генов вирулентности малой РНК PinT. Стрелками с острыми концами показано положительное влияние, стрелками с тупыми концами — негативное влияние.
Полученная информация позволяет утверждать, что PinT регулирует переход сальмонеллы из фазы вирулентности в фазу внутриклеточного выживания, оказывая влияние на гены островов SPI-1 и SPI-2.
Влияние PinT на реакцию хозяйской клетки
С помощью одновременного секвенирования РНК бактерии и клетки-хозяина удалось выяснить, как малые РНК сальмонеллы влияют на процессы внутри клеток HeLa. В эксперименте использовали Salmonella Typhimurium дикого типа и с делецией pinT.
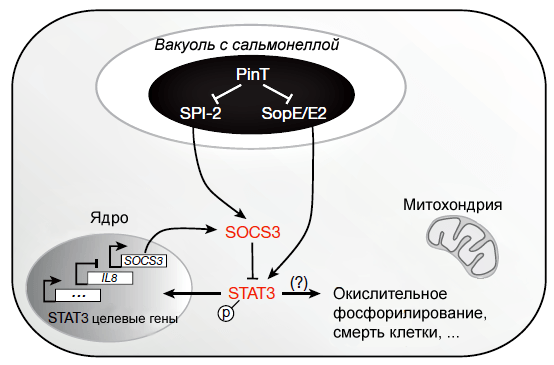
Оказалось, что в отсутствие PinT наблюдается повышенная активность регуляторного белка SOCS3, который ингибирует фосфорилирование транскрипционного фактора STAT3, запрещая его активацию и транспортировку в ядро клетки (рис. 3). SOCS3 — белок воспалительного сигнального пути, играющий важную роль в течении инфекции: небольшое воспаление понижает способность сальмонеллы конкурировать с кишечной микробиотой, а слишком сильное — приводит к уничтожению бактериальных клеток хозяином [1]. Роль PinT в данном случае — поддерживать оптимальное для развития бактерий количество SOCS3 в человеческой клетке.
Рисунок 3. Схема влияния PinT-зависимых бактериальных эффекторов на биохимические процессы в хозяйской клетке. Стрелками с острыми концами показано положительное влияние, стрелками с тупыми концами — негативное влияние.
Кроме того, делеция PinT повышала выработку интерлейкина 8 (IL-8) — провоспалительного хемокина.
То есть посредством влияния на эффекторные белки SopE и SopE2 PinT регулирует синтез интерлейкина 8, а контролируя некоторые гены острова SPI-2, оказывает влияние на активность SOCS3 [12], [13].
Вместе с тем метод dual RNA-seq показал связанный с инфекцией синтез длинной некодирующей РНК (long noncoding RNAs, lncRNAs). Определенные днРНК очень быстро отвечают на PinT-зависимые изменения в клетке, а потому могут использоваться при диагностике сальмонеллёза как чувствительные маркеры ранних стадий инфекции.
Сальмонелла также устойчиво влияет на митохондрии клетки-хозяина: бактериальный штамм без PinT вызывал гиперактивацию синтеза РНК митохондрий, включая гены пути окислительного фосфорилирования, а также изменял внутриклеточную локализацию самих митохондрий.
В дальнейшем метод высокопроизводительного двойного секвенирования РНК поможет понять многие скрытые взаимосвязанные процессы в болезнетворных бактериях и хозяйских клетках при инфицировании не только сальмонеллой, но и другими микроорганизмами.
Одноклеточное секвенирование: разделяй, изучай и властвуй
Одноклеточное секвенирование: разделяй, изучай и властвуй
Одноклеточное секвенирование дает возможность увидеть более детальную картинку. Рисунок в полном размере.
Автор
Редакторы
Современные методы полногеномного секвенирования позволяют считывать миллиарды последовательностей нуклеиновых кислот (НК) — ДНК или РНК — за считанные часы. Человечество вплотную подобралось к пониманию механизмов реализации генетического материала в живых системах, и новые технологии одноклеточного секвенирования с возможностью визуализации пространственного положения нуклеиновых кислот внутри или вне клетки, похоже, стали еще одним шагом к этой цели.
Ультрасовременные методы
Методы, которыми оперирует современная наука, постоянно совершенствуются. Некоторые области развиваются настолько стремительно, что порой даже специалистам сложно уследить за новейшими приборами и модификациями методик. Так происходит, например, в геномике. Другие же области (такие как гистология и связанные с ней световая и флуоресцентная микроскопия) развиваются куда медленнее, но всё равно расширяются и обзаводятся более высокопроизводительными методами. В статьях нового спецпроекта (он продолжает наш хит — «12 биологических методов в картинках») мы хотим рассказать, какие методы помогают ученым совершать новые открытия сегодня, и чего нам ждать в будущем.
Партнер этой статьи — «СкайДжин».
Пять причин выбрать SkyGen:
К середине прошлого столетия, в момент открытия нуклеиновых кислот (НК — ДНК и РНК) как носителей генетической информации живых организмов, перед исследователями встал вопрос о возможности прочтения их последовательности — секвенирования. Десятки лет и поступательный прогресс во многих отраслях науки и инженерии сделали возможным сначала определение последовательности отдельных фрагментов ДНК, а затем и секвенирование целых геномов.
К настоящему времени разработано и выведено на биотехнологический рынок значительное количество методов секвенирования НК. Самый популярный и надежный из них — секвенирование методом «терминаторов» — разработал британский биолог Фредерик Сэнгер с коллегами в далеком 1977 году [1]. За десятки лет данный метод неоднократно модифицировался (например, радиоактивные метки заменили флуоресцентными), однако идея, лежащая в его основе, не изменилась. Анализируемый, предварительно многократно амплифицированный фрагмент ДНК (до 1000 пар нуклеотидов (п.н.) — букв генетического алфавита) используется как матрица для синтеза комплементарной (дочерней) цепочки ферментом ДНК-полимеразой в присутствии дезоксинуклеозидтрифосфатов (А, Т, Г, Ц — аденина, тимина, гуанина и цитозина). Кроме того, в смеси находятся флуоресцентно меченные дидезоксинуклеозидтрифосфаты, которые прерывают реакцию синтеза из-за особенности химического строения. Благодаря флуоресценции и электрофорезу в геле можно точно установить длину конкретного секвенируемого фрагмента ДНК и последнюю букву в нем. Поскольку в реакционной смеси параллельно проходят тысячи реакций, используя этот уже полностью автоматизированный метод, можно восстановить последовательности десятков ДНК-фрагментов.
Несмотря на то, что определение последовательностей НК по Сэнгеру до сих пор является «золотым» стандартом секвенирования и нашло свое применение в различных жизненно важных отраслях (таких как медицинская генетика и криминалистика), его производительность оставляет желать лучшего [2], [3].
Геномы многих растений и позвоночных животных, в том числе и человека, состоят из миллиардов «букв», и определение их полных последовательностей с использованием настолько «олдскульных» методов экономически нецелесообразно.
За последние годы сразу несколько технологий глубокого секвенирования внедрились в современную биологическую науку. Некоторые из них — лигазное секвенирование или пиросеквенирование, о которых можно почитать в нашей обзорной статье «12 методов в картинках: секвенирование нуклеиновых кислот» [2], — уже не поддерживаются их разработчиками, в то время как секвенирование на молекулярных кластерах (также известное как технология Illumina), или одномолекулярное секвенирование в реальном времени (чуть позднее к ним присоединилась технология cPAS секвенирования), прочно заняли свои ниши. Одна часть этих технологий прекрасно подходит для анализа новых, ранее не опубликованных геномов или поиска длинных некодирующих РНК, тогда как другая эффективна и обходится дешевле при ресеквенировании или анализе экспрессии генов. Более того, исследователи могут комбинировать разнообразные методы для достижения наиболее интересных результатов. В частности, одномолекулярное секвенирование — которое осуществляется благодаря приборам PacBio, — также как и нанопоровое секвенирование (Oxford Nanopore) [2], [10], регулярно используется вместе с технологией Illumina для хромосомной сборки новых геномов, в том числе и полиплоидных [11–13].
Одноклеточное секвенирование (ОКС, scRNA-seq)
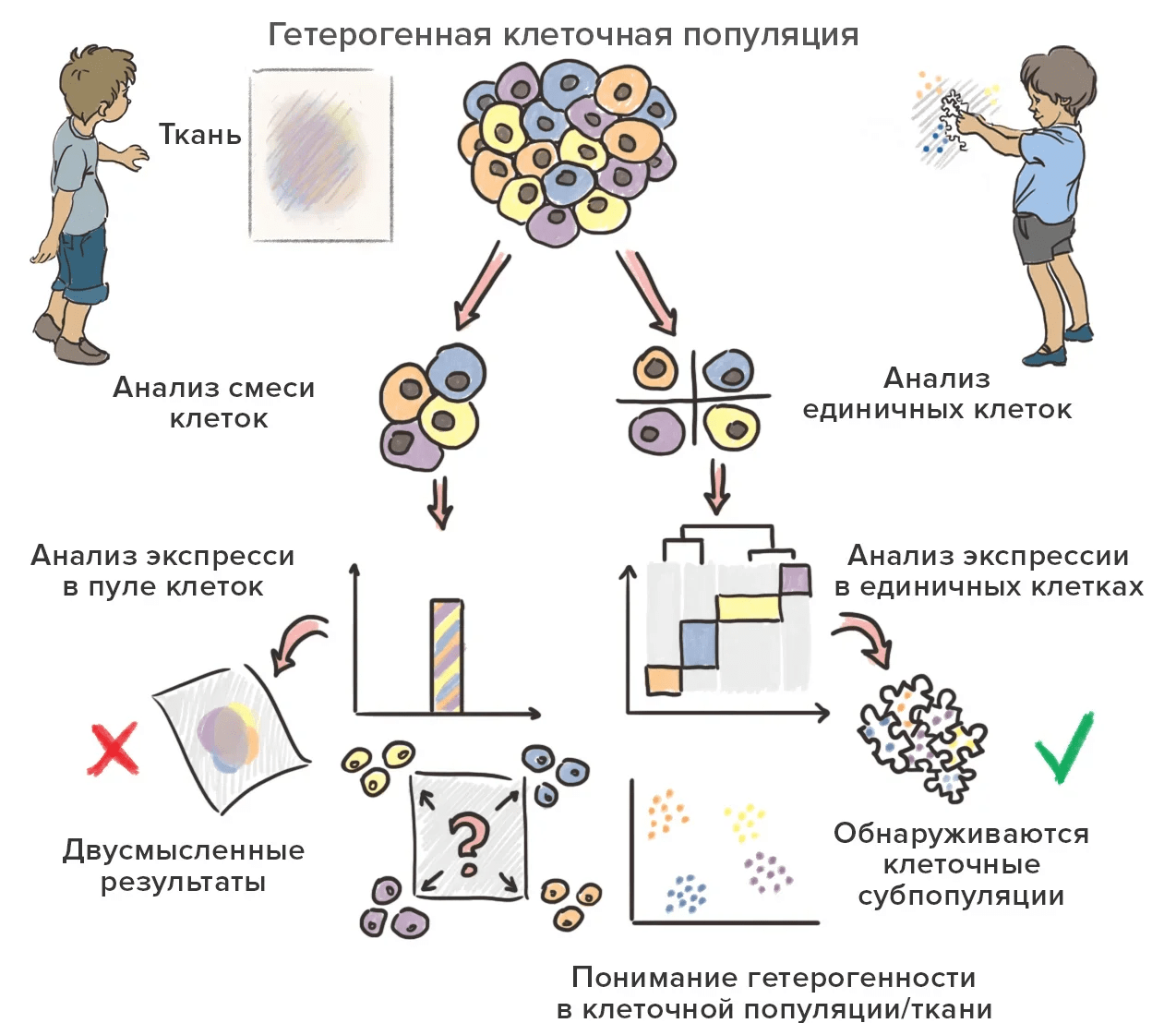
Таким образом, на сегодняшний день секвенирование ДНК и РНК — это во многом рутинная процедура, которая служит лишь в качестве одного из множества методов современной молекулярной биологии и биотехнологии. Используемые сегодня методы секвенирования НК позволяют оценивать не только последовательность ДНК, но и экспрессию кодируемых ею молекул РНК (транскриптом), а также разнообразные типы модификаций генома и транскриптома, такие как метилирование, тесно связанное с активностью генов. В то же время перед учеными то и дело встают новые задачи. Так, несмотря на генетическую идентичность клеток в одном организме, иногда требуется оценить разницу между одиночными клетками и их субпопуляциями; более того, последние исследования показали, что из ста триллионов клеток, составляющих человеческое тело, не найдется и двух с полностью одинаковым геномом — из-за мутаций, накапливающихся в эмбриогенезе и в течение жизни организма [14].
Дело в том, что большинство широко применяемых методов анализа нуклеиновых кислот позволяет работать с тканями, которые состоят из абсолютно разных клеточных субтипов. При этом, анализируя образцы ДНК или РНК из таких экстрактов, ученые получают некие усредненные результаты и редко могут прицельно дотянуться до конкретной функции или механизма.
В результате исследователи, обращаясь к классическим методам глубокого секвенирования, нередко сталкиваются с трудностями — например, при анализе геномов некультивируемых микроорганизмов, изучении генетического мозаицизма [14] или геномном анализе клеточных субпопуляций раковых опухолей. Связаны эти трудности, как правило, с биоинформатическим анализом данных, а иногда эксперименты вовсе не дают необходимых результатов [15]. Не меньше проблем у ученых до недавнего времени возникало в ходе анализа экспрессии генов или метилирования в комплексных тканях, состоящих из разных типов клеток (например, тканях головного мозга), или в тканях, пронизанных кровеносными сосудами [16], [17].
Ответом на все эти вызовы стали технологии одноклеточного секвенирования (ОКС). Они позволяют получать информацию о многомерности тканей, выявляя различия в клеточных популяциях, и оценивать клеточные взаимосвязи, а не получать лишь среднее значение для многих клеток, теряя информацию об их гетерогенности, как это было раньше. И если еще недавно широкое распространение ОКС сдерживалось его высокой стоимостью, то по мере появления новых методов пробоподготовки и секвенирования сегодня данный подход используется всё чаще [18].
Одноклеточное секвенирование можно разделить на четыре важнейших этапа:
На протяжении всех четырех этапов важно обращать внимание на ряд экспериментальных параметров и критических точек, следование которым позволит провести успешный эксперимент и получить достоверные данные [19].
Пожалуй, одним из важнейших этапов при одноклеточном секвенировании является пробоподготовка, поскольку она может в значительной мере повлиять на результат. Например, ткани с высокими показателями адгезии между клетками должны быть тщательно диссоциированы до одноклеточного уровня. С другой стороны, экспериментатор должен отдавать себе отчет в том, что более разрушительные методы суспендирования клеток могут иметь фатальные последствия для результатов, приводя к гибели части клеток и тем самым нарушая соотношения субпопуляций в ткани. На сегодняшний день для диссоциации тканей используют три основных подхода: ферментативный (обработка биологического образца ферментами, разрушающими межклеточный матрикс, — коллагеназой, эластазой, трипсином и другими), механический (диссекция клеток, пипетирование, центрифугирование и др.), а также комбинированный, который включает в себя первые два.
Кроме того, в некоторых случаях необходима не только диссоциация клеток и приготовление их суспензий, но и «обогащение» редко представленных клеточных субпопуляций в общем клеточном пуле, а также элиминация мертвых клеток. Для этих случаев может быть использована целая плеяда методов, которые позволяют идентифицировать определенную субпопуляцию клеток, основываясь на их массе, размере, плотности. Различные флуоресцентные метки, в свою очередь, применяются для идентификации и последующей сортировки определенных типов клеток [20].
Широкое распространение при подготовке биологических образцов к ОКС получили методы проточной цитофлуориметрии. Проточная цитометрия является ценным инструментом для контроля качества, поскольку с ее помощью одновременно можно оценивать множество показателей, включая размер клеток, их жизнеспособность, а также адгезию между клетками. Подробнее о методах цитометрии можно почитать в ранее опубликованной на «Биомолекуле» статье «12 методов в картинках: проточная цитофлуориметрия» [21].
Chromium Controller: как анализировать множество клеток сразу
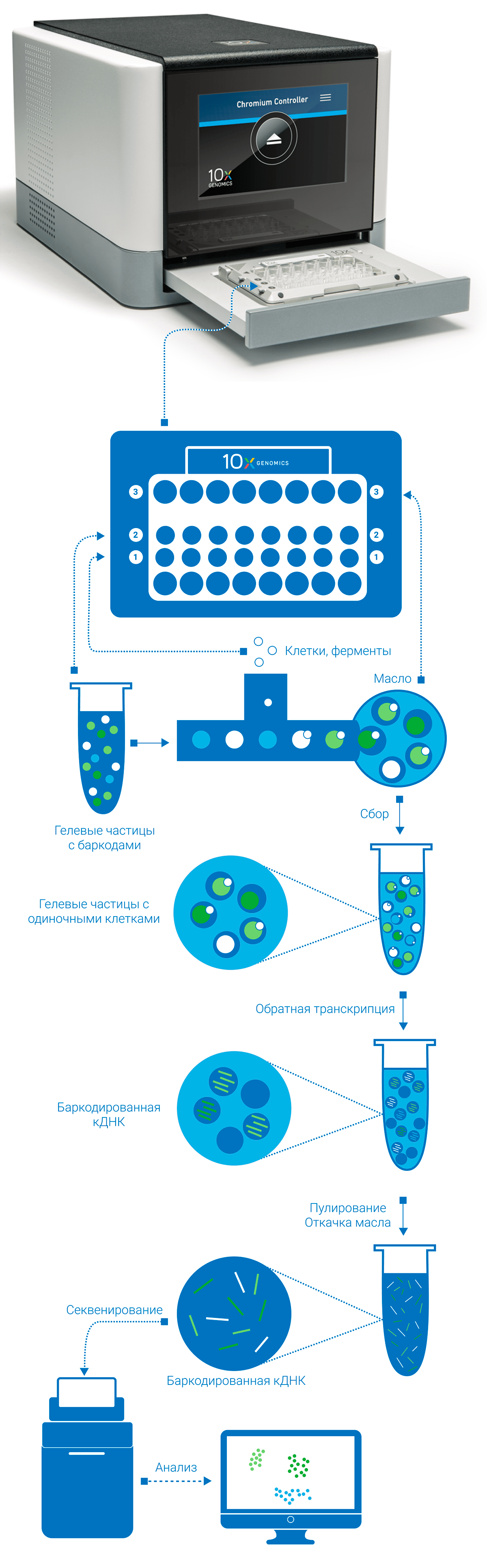
Прибор Chromium Controller от компании 10x Genomics способен быстро разделять клетки по отдельным каплям и баркодировать их ДНК, что позволяет проводить параллельный высокопроизводительный анализ десятков тысяч клеток.
Рисунок 1. Схема работы прибора Chromium Controller для секвенирования ДНК отдельных клеток или ядер
Прибор компактен и очень прост в использовании: всё, что требуется пользователю — это правильно загрузить чип и поместить его в прибор. В чип вносят клетки, реагенты (гелевые частицы) и масло (рис. 1).
Весь эксперимент можно разделить на пять этапов:
Материал предоставлен партнером — компанией «СкайДжин»
После лизиса из каждой клетки выделяется генетический материал, используемый для приготовления ДНК-библиотек. Поскольку каждая клетка содержит лишь один диплоидный геном, его необходимо предварительно амплифицировать, так как современные платформы для секвенирования не позволяют работать с одной или двумя копиями генома (рис. 2). Для наработки достаточного количества копий при пробоподготовке используют разнообразные методы полногеномной амплификации (whole genome amplification, WGA), позволяющие избежать значительного количества ошибок, — например, изотермическую ПЦР. В случае работы с различными типами РНК пул транскриптов предварительно переводят в комплементарную ДНК (кДНК) при помощи обратной транскриптазы, а затем также амплифицируют отдельно для каждой клетки. В настоящее время особой популярностью по-прежнему пользуется метод амплификации со множественным замещением цепи (multiple displacement amplification, MDA) с применением полимеразы phi29 (выделенной из одноименного бактериофага). Данная полимераза славится своей высокой процессивностью, малым количеством ошибок и широтой покрытия генома (около 84%) [24].
Другой метод полногеномной амплификации, известный под аббревиатурой MALBAC (Multiple annealing and looping-based amplification cycles), объединяет в себе MDA и обычную ПЦР и дает возможность не только амплифицировать значительную часть генома (более 50%) или транскриптома, но и добиться высокой равномерности покрытия и меньшей частоты потери аллелей относительно использования полимеразы phi29 [24] — она предпочтительно амплифицирует один аллель в гетерозиготном состоянии.
Таким образом, MDA и MALBAC имеют как преимущества, так и недостатки, а поэтому должны применяться в зависимости от целей эксперимента.
Рисунок 2. Основные этапы подготовки образцов ДНК и РНК для одноклеточного секвенирования
После выделения и амплификации НК для каждой анализируемой клетки исследователь должен выбрать дальнейшее направление пробоподготовки, которая в будущем определит методику секвенирования. Сегодня на биотехнологическом рынке представлены самые разные технологии определения последовательности НК, способные решать стоящие перед исследователем задачи. Вот главные из них:
Первые четыре технологии подробно рассмотрены ранее на «Биомолекуле» («12 методов в картинках: секвенирование нуклеиновых кислот» [2] и «Нанопоровое секвенирование: на пороге третьей геномной революции» [10]), тогда как технология сPAS, выведенная на рынок коммерческих приборов китайской компанией BGI Genomics, вошла в повседневный обиход лишь несколько лет назад, но уже слегка потеснила технологию массового секвенирования, предлагаемую компанией Illumina.
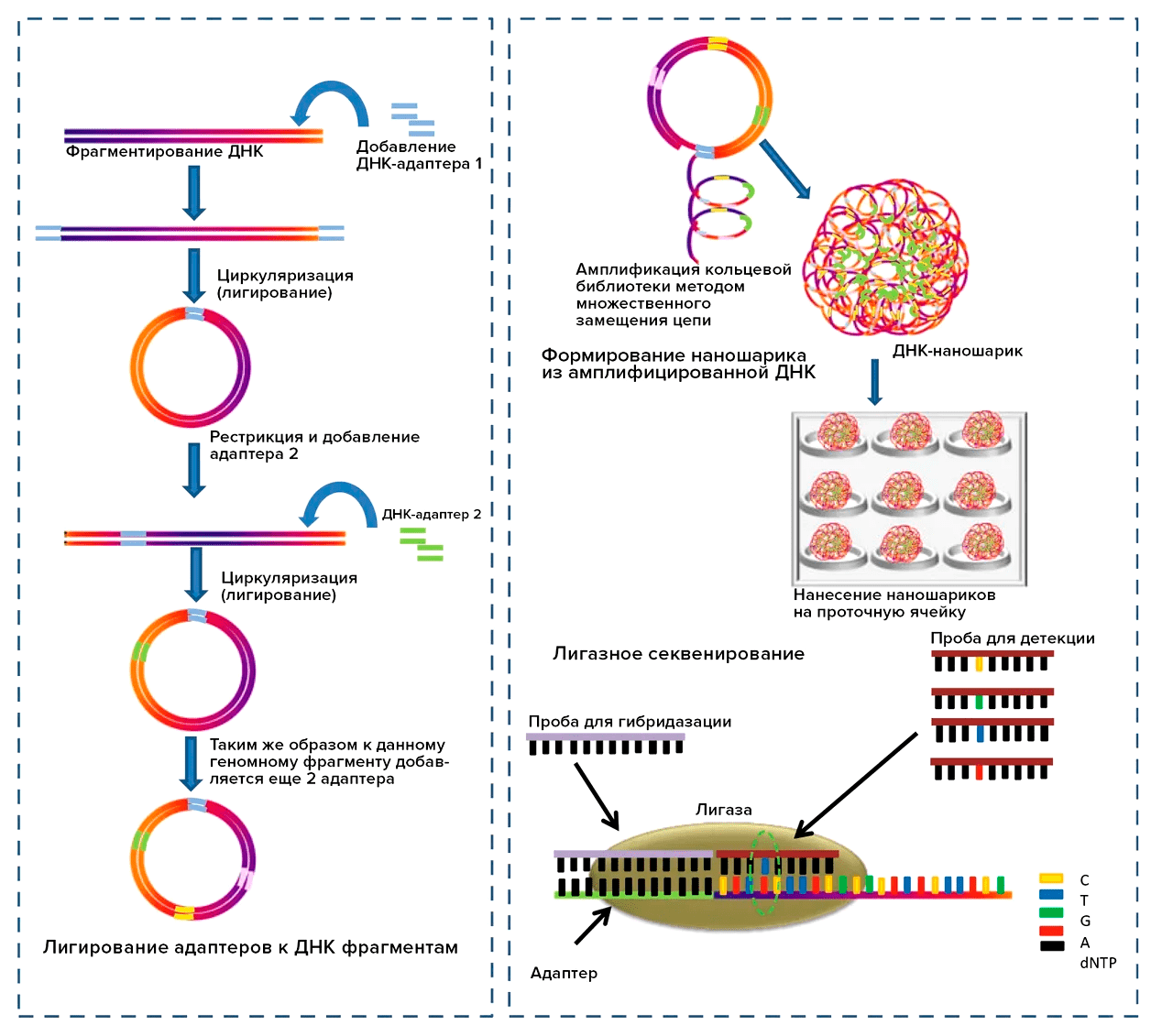
На примере работы с геномной ДНК суть метода cPAS (рис. 3) заключается в следующем. К обоим концам предварительно фрагментированной ДНК (около 500 пар оснований) лигируют адаптеры. Затем полученную библиотеку циркуляризируют и амплифицируют, используя ранее упомянутый метод с множественным замещением цепи (MDA) по принципу катящегося кольца, то есть исходный фрагмент прочитывается многократно. Такой подход позволяет избежать клонального накопления ошибок полимеразы. После того, как количество амплифицированных фрагментов достигнет нескольких сотен, они укладываются с использованием запатентованной технологии в ДНК-наношарик (DNA nanoball, DNB) [25].
Рисунок 3. Технология cPAS-секвенирования. Фрагментированная геномная ДНК лигируется с четырьмя адаптерами, формируя кольцевую ДНК-библиотеку. ДНК-наношарик формируется путем репликации по принципу катящегося кольца. Каждый ДНК-наношарик помещается в определенное место на ячейке секвенатора. Считывание ДНК осуществляется с использованием принципов гибридизации и лигазного секвенирования.
Полученные ДНК-фрагменты, компактизированные в виде ДНК-наношариков, наносятся на проточную ячейку секвенатора, где закрепляются за счет электростатических сил. На проточной ячейке проводят циклический процесс секвенирования, в котором объединяются принципы гибридизации и лигазного секвенирования. Реакционная смесь для синтеза комплементарной ДНК подается на поверхность проточной ячейки и содержит ферменты, а также четыре типа флуоресцентно меченых проб, состоящих из нуклеотидов универсальной специфичности (способных связаться с любым из нуклеотидов матричной ДНК), а также одного известного нуклеотида (A, T, G или С) на 5′-конце. После гибридизации пробы и специфичного нуклеотида лигирования по 5′-концу камерой с ПЗС-матрицей считывается флуоресценция пробы, а затем универсальные нуклеотиды и флуоресцентный краситель смываются. Эта серия шагов продолжается определенное количество раз, число которых задает пользователь [25].
Биоинформатический анализ данных одноклеточного секвенирования
Биоинформатический анализ данных одноклеточного секвенирования, несмотря на идеологическое сходство с классическим, рутинным анализом транскриптома в тканях и клеточных культурах (подробная информация представлена в классическом обзоре: A survey of best practices for RNA-seq data analysis [26]), представляет собой не самую тривиальную часть эксперимента, и в рамках данного обзора крайне сложно пройтись по всем подводным камням, которые ожидают компьютерного биолога. Уж слишком много приложений одноклеточного секвенирования существует сейчас, да и каждое конкретное исследование, ткань или модель требуют особого подхода.
Важный момент при анализе данных одноклеточного секвенирования — необходимость нормализации изучаемых образцов. Дело в том, что в ходе экспериментальных процедур (сепарация клеток, выделение НК, полногеномная амплификация и создание библиотек) существует значительная вероятность ошибок. Эти, казалось бы, незначительные этапы в дальнейшем могут существенно влиять на ширину и глубину покрытия генома/транскриптома, на смещение в числе определенных типов клеток и их субпопуляций, а также на соотношение аллелей. Данные проблемы решаются воспроизводимостью экспериментальных процедур, добавлением технических и биологических повторностей, а также использованием дополнительных математических методов [19].
При геномном секвенировании глубина покрытия отдельных клеток значительно меньше, чем в массовых экспериментах. Таким образом, увеличение разрешения до клеточного уровня с использованием технологий ОКС означает также снижение достоверности обнаруживаемых механизмов, которые становятся более стабильными, когда суммируются (например, при анализе ткани). В конечном счете, задачи, которые ранее казались рутинными (например, поиск единичных нуклеотидных вариаций или геномных перестроек в субпопуляциях опухоли), требуют значительной методологической осторожности при обращении с данными одноклеточного секвенирования [27].
Так же как и при геномном секвенировании, анализ транскриптомных данных ОКС теоритически позволяет получить полное представление о взаимодействии транскриптов внутри отдельных клеток. Однако анализ экспрессии генов единичных клеток зачастую страдает от недостаточного покрытия, что в результате объединяет два различных типа нулевых значений в экспрессии конкретного транскрипта:
Во многом результаты транскриптомных исследований на одноклеточном уровне (впрочем, как и геномных) напрямую зависят от лабораторной части эксперимента, используемой платформы для пробоподготовки и секвенирования, глубины секвенирования, уровня экспрессии конкретного гена и так далее [27].
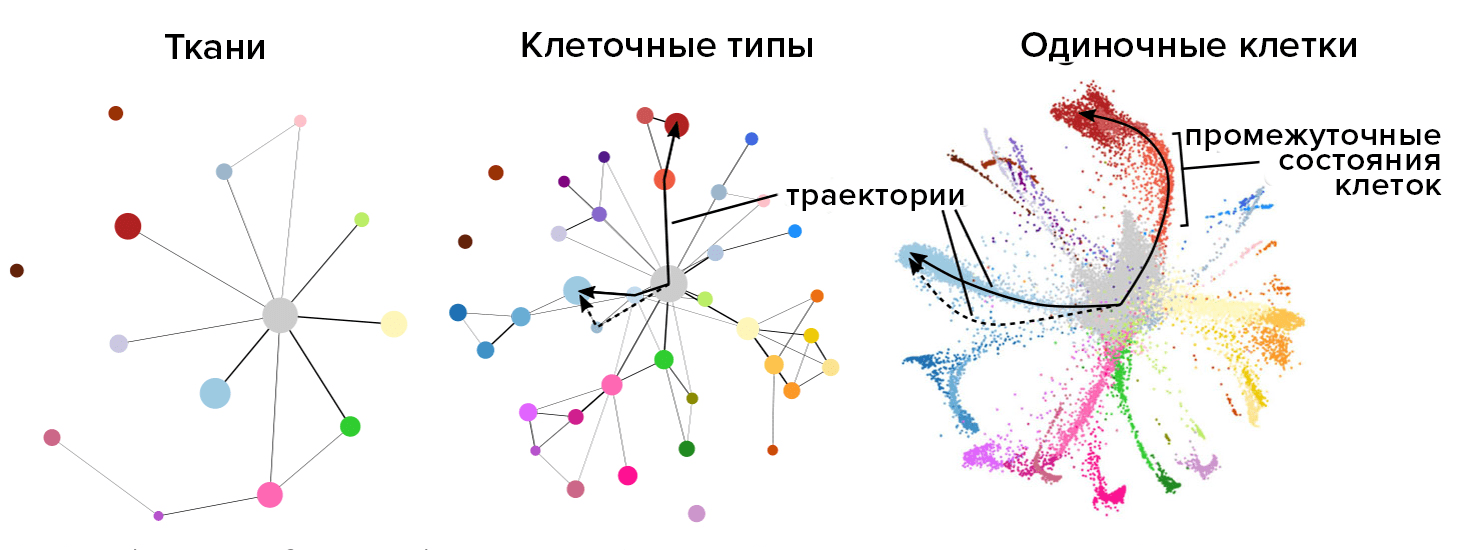
Подводя итог, можно сказать, что одноклеточное секвенирование НК может выявить сложные и редкие популяции клеток, найти регуляторные взаимосвязи между генами и отследить траектории развития отдельных клеточных линий. Дальнейшие технические усовершенствования на уровне молекулярной и клеточной биологии и доступных биоинформатических инструментов значительно облегчат как фундаментальные исследования, так и прикладное применение (медицина и биотехнология) этих технологий секвенирования [19].
Пространственная транскриптомика: новый шаг вглубь клетки и её машинерии
Распознавание пространственно-временных паттернов экспрессии генов имеет решающее значение для понимания основных биологических принципов от эмбриогенеза до возникновения и протекания болезней. Новые методы пространственной транскриптомики в единичных клетках расширяют наши знания о молекулярных процессах, протекающих внутри клеток и во внеклеточной среде. Эти подходы позволяют, например, реконструировать в цифровом виде паттерны эмбриональной экспрессии в 3D и успешно идентифицировать новые домены экспрессии, типы клеток и особенности тканей. Такие технологии открывают путь к беспристрастному и исчерпывающему анализу уровней экспрессии генов в пространственном и количественном выражении, способствуют пониманию функционирования генома и эпигенома в клетке.
Подробнее о роли микроокружения в прогрессировании онкологических заболеваний можно почитать здесь: «Опухолевые разговоры, или Роль микроокружения в развитии рака» [22].
Современные методы молекулярной биологии, флуоресцентной гибридизации in situ (FISH) и микроскопии позволили не только анализировать синтез молекул РНК из отдельных тканей или отдельных клеток, но и визуализировать их в живых системах. Подобная визуализация in vivo может быть осуществлена посредством гибридизации меченого зонда, комплементарного транскриптам-мишеням. Более того, современные системы считывания анализа флуоресценции (например, сложные зонды, использующие комбинации флуорофоров) позволяют избегать спектрального перекрытия различных меток и тем самым анализировать значительное количество мишеней в клетке. В то же время этот подход не подходит для идентификации положения всех транскриптов в клетке [31].
Общей проблемой методов, основанных на FISH, считается их высокая цена и связанная с ней ограниченность в выборе транскриптов, а также высокий уровень фона аутофлуоресценции в непрозрачных образцах тканей [30].
Методы флуоресцентной гибридизации in situ, как показала практика, имеют существенные ограничения по числу реализующихся транскриптов, которые возможно описать. Поэтому были предложены другие методы, основанные на идентификации положения транскриптов в клетке, их захвата и последующего секвенирования ex situ. Такая концепция реализована в нескольких методологических подходах, где возможен анализ всего транскриптома. Однако основным препятствием для продвижения этих методов долгое время являлась ограниченная эффективность захвата РНК, которая становится всё более сложной при более высоком разрешении.
Первый способ пространственной транскриптомики был опубликован в 2016 году и в настоящее время коммерциализирован компанией 10x Genomics под названием 10x Visium. Суть данной методики заключается в приготовлении тонких (10–50 мкм) гистологических срезов и нанесении их на специальные предметные стекла с иммобилизованными на них поли-dT-олигонуклеотидами, для каждого из которых известно местоположение по Х- и Y-осям, и каждый из которых имеет специфический баркод. В дальнейшем ткань фиксируется, окрашивается и визуализируется (рис. 4).
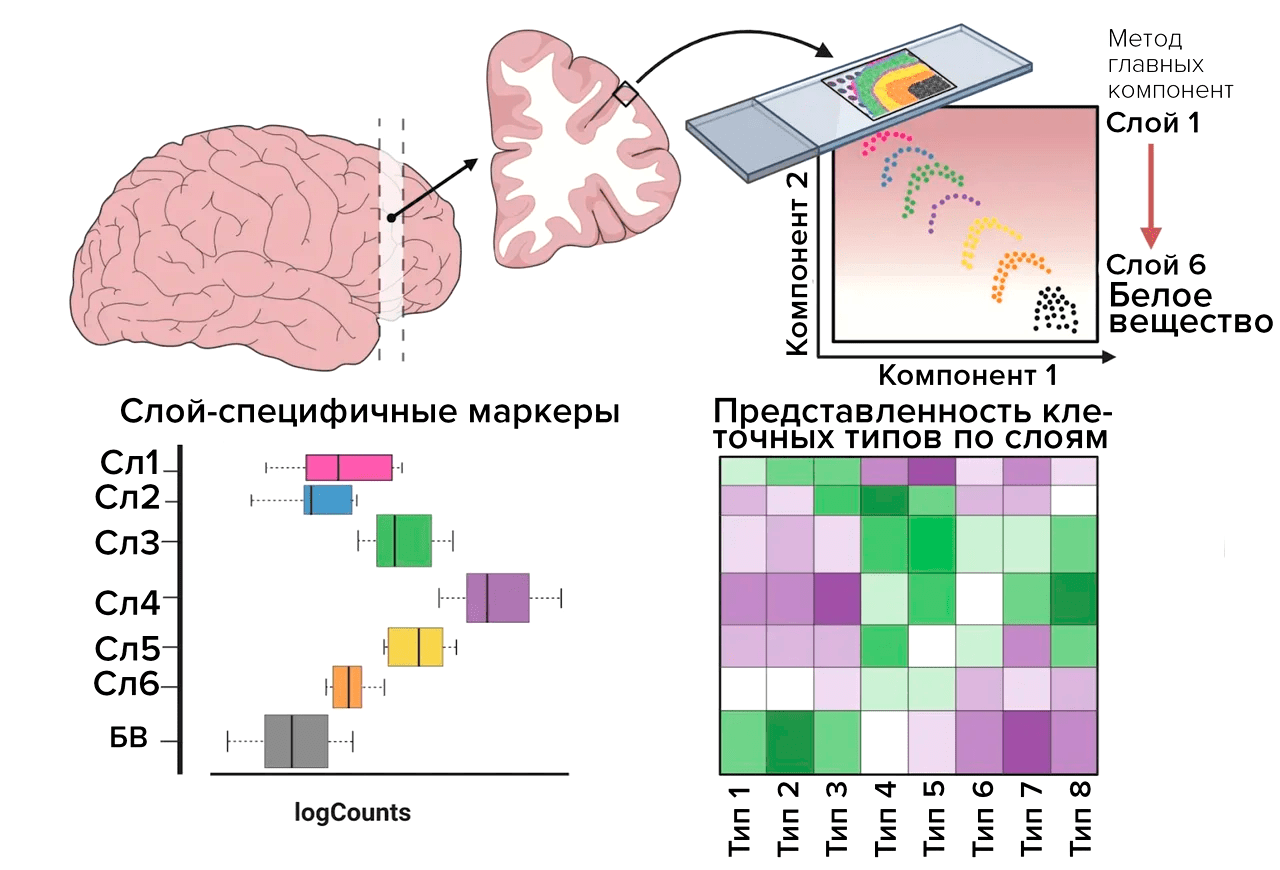
Рисунок 4. Пример использования пространственной транскриптомики при анализе экспрессии генов в тканях префронтальной коры головного мозга человека. С помощью набора реагентов для пермеабилизации происходит снижение проницаемости клеточных мембран, и информационные РНК (матричные РНК, мРНК) диффундируют вниз к твердой поверхности слайда, локально гибридизуясь с иммобилизованными на его поверхности поли-dT-олигонуклеотидами. Затем методом обратной транскрипции проводится синтез кДНК и другие этапы подготовки библиотеки для последующего секвенирования с использованием одной из популярных платформ. Данные в дальнейшем экстраполируются на изображение среза ткани, полученное в начале эксперимента, предоставляя тем самым информацию о расположении конкретного транскрипта в клетке [32].
Области применения технологий single cell sequencing от компании 10x Genomics
Анализ уровня экспрессии единичных клеток, как и анализ пространственной экспрессии генов, может применяться для исследований в различных областях, включая:
Нейробиология
Нервная система — это сложная сеть, состоящая из различных типов клеток, которые имеют множество сигнальных путей и синаптических взаимодействий. При нарушении функций или взаимодействий компонентов центральной нервной системы (ЦНС) возникают нейродегенеративные заболевания, причем их причиной может послужить как травма, так и заболевание или генетическая вариативность (рис. 5).
Рисунок 5. Нейробиология — широкое поле для исследований
Традиционные методы изучения клеточных типов и подтипов в нервной ткани — это иммуногистохимия, гибридизация in situ, анализ на микрочипах. Эти технологии основаны на детекции небольшого количества известных мишеней, таких как клеточные маркеры, миелинизированные аксоны или амилоидные бляшки, и требуют большого количества образца для анализа. Также эти методы не могут предоставить информацию о гетерогенности клеточных субтипов и их уровней экспрессии.
В то же время анализ на уровне единичных клеток позволяет:
Онкология
Несмотря на огромное количество исследований в области онкологии, у ученых всё еще остается множество вопросов. Какие механизмы лежат в основе возникновения и прогрессии опухоли? Как различная реакция на терапию может быть применена к разным пациентам с различными типами опухолей? Как идет метастазирование в зависимости от типа опухоли и особенностей больного? Ответить на эти и многие другие вопросы может анализ на уровне единичных клеток. 10x Genomics разработала ряд готовых протоколов для различных задач:
Иммунология
На фронте многих важных научных и медицинских достижений находятся исследования в области иммунологии, такие как профилирование новых патогенов, разработка вакцин, разработка иммуноонкологических методов лечения, изучение аутоиммунных заболеваний, подбор тканей для безопасной трансплантации органов и многое другое. Область действительно масштабная по своим направлениям, ниже представлены некоторые из них с точки зрения анализа на уровне единичных клеток:
Инфекционные заболевания. Узнайте, как патогены заражают клетки-хозяева и вызывают иммунные реакции. Свяжите иммунный ответ хозяина с выздоровлением или тяжестью инфекции.
Аутоиммунные реакции. Расшифруйте основные механизмы неправильно направленных иммунных реакций. Исследуйте патофизиологию заболеваний в отдельных клетках и системах органов.
Трансплантология. Контролируйте клиническое ведение трансплантации органов или гематологических стволовых клеток. Изучите иммунологическую основу заболеваний, связанных с трансплантацией.
Поиск новых лекарств. Определите репертуар рецепторов и антигенную специфичность адаптивных иммунных клеток. Ведите разработку вакцин, выявляйте профилактические антитела и проводите целенаправленную иммунотерапию.
Клеточная и молекулярная иммунология. Ведите фундаментальные исследования иммунной системы в области здоровья и болезней. Характеризуйте разнообразие, функции и организацию иммунных клеток в организме.
Аллергии и воспаление. Исследуйте особенности врожденной и адаптивной иммунной активации. Расшифруйте механизмы иммунной гиперактивации в ответ на аллерген или травму.
Иммуноонкология. Определите инфильтрирующие иммунные клетки в микроокружении опухоли. Охарактеризуйте функции иммунных клеток и репертуар рецепторов.
Материал предоставлен партнером — компанией «СкайДжин»
Кроме описанного выше, существуют и другие методы пространственного секвенирования. Один из них (Slide-Seq) использует магнитные шарики с иммобилизованными поли-dT-олигонуклеотидами, на которых проходит считывание. Шарики случайно наносятся на слайд, и их местоположение декодируется перед началом нанесения на этот слайд образца [33]. Не менее интересной выглядит технология GeoMx, которая уже показала свою эффективность при работе со сложными образцами из парафиновых блоков. Кроме анализа экспрессии генов платформа GeoMx Digital Spatial Profiling (DSP) также предоставляет возможность пространственного профилирования белков. В процессе работы пользователь вручную выбирает интересующие его области клетки с помощью микроскопии. Затем эти области обрабатываются УФ-светом, вызывая высвобождение либо РНК-зонда связанного с транскриптом-мишенью, либо белка, помеченного антителом [34].
Биоинформатический анализ данных при пространственном транскриптомном анализе
Клетки разных типов пространственно и структурно организованы в тканях и выполняют свои разнообразные функции. Расшифровка сложной пространственной архитектуры гетерогенной ткани важна для понимания клеточных механизмов и функций в норме и при различных заболеваниях. Быстрое развитие технологий scRNA-seq позволило начать крупномасштабные исследования гетерогенных клеток и работ по отслеживанию родственных связей внутри ткани. К сожалению, из-за отсутствия пространственной информации scRNA-seq не способен идентифицировать структурную организацию гетерогенных клеток в сложной ткани. Поэтому в качестве дополнения к scRNA-seq были введены методы пространственного профилирования транскриптома. Чтобы выявить пространственную цитоархитектуру в тканях, используют описанные выше технологии, позволяющие проводить количественный анализ транскриптома с пространственным разрешением в отдельных срезах ткани. Технологические новшества и новые подходы в пространственной транскриптомике открыли неизведанный ландшафт, где транскрипционная информация помещается в пространственный контекст. Кластеризация — центральный компонент при анализе данных этого типа. Однако выбор количества кластеров для использования и интерпретация их взаимосвязей могут быть затруднительны [35], [36].
Новые технологии пространственной транскриптомики могут пространственно индексировать транскрипты и измерять профили экспрессии, улучшая наше понимание точной архитектуры тканей. Однако разрешение данных пространственной транскриптомики намного ниже, чем уровень одиночной клетки. Транскрипты, захваченные в определенном месте, обычно представляют смесь из 10–20 клеток. В результате измеренные экспрессии генов отражают хоть и небольшую, но смесь клеток. Следовательно, выявление клеточных составов в каждой точке данных ST имеет решающее значение для исследования молекулярной и клеточной архитектуры ткани с высоким разрешением [36].
К настоящему времени для кластеризации и визуализации данных разработано не так много программных продуктов, хотя определенные успехи в этой области уже появляются, в частности, с использованием искусственного интеллекта и математических моделей удалось определить расположение клеток и транскриптов, синтезируемых в них (рис. 6) [36].
Рисунок 6. Различные уровни разрешения и различные результаты, получаемые в зависимости от использованных методов
Заключение
С появлением NGS-технологий в начале века человечество получило ключи и новые возможности для понимания молекулярных процессов, управляющих жизнью, однако анализ геномов, транскриптомов или эпигеномов представлялся серьезной проблемой при анализе комплексных тканей (кровь, нервная ткань) и органов, состоящих из различных субпопуляций клеток, различающихся по своему функционалу, а значит, и по тем генам, которые в них задействованы. Новые технологии одноклеточного секвенирования и пространственной транскриптомики придают новый импульс развитию современной геномики [18].
Технологии одноклеточного секвенирования дают уникальную возможность изучать молекулярную активность отдельных нервных клеток, визуализируя нейроны во времени и пространстве. Это позволяет изучать эволюцию головного мозга [37], идентифицировать различные типы нейронов и связанных с ними клеток в мозге. На сегодняшний день в головном мозге обнаружено большое число молекулярных типов нейронов, подтипов глиальных клеток [38], [39]. Ожидается, что понимание их работы на молекулярном уровне приблизит врачей к пониманию причины возникновения ряда заболеваний головного мозга, таких как шизофрения [40], болезни Альцгеймера [41] и Паркинсона [42].
Немаловажно использование технологий одноклеточного секвенирования в анализе причин и механизмов течения ряда генетически обусловленных заболеваний человека, в частности, онкологических. Исследования последних лет показали, что генетические или геномные вариации могут приводить к образованию клеток с различными характеристиками в опухолевой ткани, что делает опухолевую ткань очень гетерогенной. Такая высокая степень гетерогенности может быть связана с механизмами онкогенеза и метастазирования, поэтому исследования клеточных субпопуляций, населяющих опухоль, крайне важны [48]. Традиционные методы секвенирования позволяют получать среднее значение сигналов в опухолевой ткани, и генетическая неоднородность опухолевых клеток часто маскируется генетическим материалом наиболее представленной субпопуляции клеток опухоли. Технологии одноклеточного секвенирования могут прекрасно компенсировать недостатки традиционных методов расшифровки генома. Клеточная карта опухолевых клеток может быть построена путем обнаружения гетерогенности опухолевых клеток и пространственной транскриптомики. Технологии ОКС уже сейчас используются при исследовании различных опухолей и имеют большое значение для разработки новых методов диагностики и терапии [48], [49].
Анализ геномного разнообразия прокариот для комплексных образцов — также одна из основных мишеней одноклеточного секвенирования из-за сложности культивирования большинства микроорганизмов. Геномика единичных клеток — это мощный способ получения последовательностей геномов прокариот в отрыве от разработки сложных способов культивирования в лаборатории. Этот подход широко применяется к морским, почвенным, подземным, организменным и другим типам микробиомов для решения широкого круга вопросов, связанных с экологией и эволюцией микроорганизмов, здоровьем человека и потенциальным применением в биотехнологии [50].